Mol:FL5FECGS0030
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 37 40 0 0 0 0 0 0 0 0999 V2000 | + | 37 40 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.7853 0.1638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7853 0.1638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7853 -0.4786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7853 -0.4786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2290 -0.7997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2290 -0.7997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3273 -0.4786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3273 -0.4786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3273 0.1638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3273 0.1638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2290 0.4850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2290 0.4850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8836 -0.7997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8836 -0.7997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4399 -0.4786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4399 -0.4786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4399 0.1638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4399 0.1638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8836 0.4850 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8836 0.4850 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8836 -1.3006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8836 -1.3006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9960 0.4849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9960 0.4849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5630 0.1575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5630 0.1575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1300 0.4849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1300 0.4849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1300 1.1396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1300 1.1396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5630 1.4669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5630 1.4669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9960 1.1396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9960 1.1396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2290 -1.4419 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2290 -1.4419 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1300 1.1396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1300 1.1396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3414 -0.7996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3414 -0.7996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2299 -1.1272 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -3.2299 -1.1272 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -2.8030 -1.6908 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.8030 -1.6908 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.1882 -1.4517 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.1882 -1.4517 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.5950 -1.4453 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.5950 -1.4453 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.0261 -1.0141 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0261 -1.0141 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6541 -1.2396 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -2.6541 -1.2396 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -3.7789 -1.0791 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7789 -1.0791 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4686 -1.7232 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4686 -1.7232 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8360 -2.0430 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8360 -2.0430 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8893 -0.5138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8893 -0.5138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8673 -0.3051 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8673 -0.3051 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8464 2.0430 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8464 2.0430 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2878 2.9403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2878 2.9403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3059 -0.9786 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3059 -0.9786 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1719 -1.4787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1719 -1.4787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1425 0.7826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1425 0.7826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6424 1.6487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6424 1.6487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 2 20 1 0 0 0 0 | + | 2 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 20 1 0 0 0 0 | + | 24 20 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 16 32 1 0 0 0 0 | + | 16 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 8 34 1 0 0 0 0 | + | 8 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 1 36 1 0 0 0 0 | + | 1 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 30 31 | + | M SAL 4 2 30 31 |
| − | M SBL 4 1 33 | + | M SBL 4 1 33 |
| − | M SMT 4 CH2OH | + | M SMT 4 CH2OH |
| − | M SVB 4 33 -2.8893 -0.5138 | + | M SVB 4 33 -2.8893 -0.5138 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 36 37 | + | M SAL 3 2 36 37 |
| − | M SBL 3 1 39 | + | M SBL 3 1 39 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 39 -1.1425 0.7826 | + | M SVB 3 39 -1.1425 0.7826 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 34 35 | + | M SAL 2 2 34 35 |
| − | M SBL 2 1 37 | + | M SBL 2 1 37 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 37 1.6388 -0.6848 | + | M SVB 2 37 1.6388 -0.6848 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 32 33 | + | M SAL 1 2 32 33 |
| − | M SBL 1 1 35 | + | M SBL 1 1 35 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 35 2.8464 2.043 | + | M SVB 1 35 2.8464 2.043 |
| − | S SKP 8 | + | S SKP 8 |
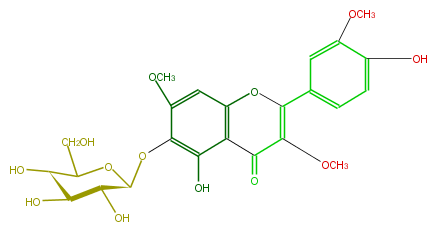
| − | ID FL5FECGS0030 | + | ID FL5FECGS0030 |
| − | KNApSAcK_ID C00005676 | + | KNApSAcK_ID C00005676 |
| − | NAME Chrysosplenoside C | + | NAME Chrysosplenoside C |
| − | CAS_RN 24274-45-1 | + | CAS_RN 24274-45-1 |
| − | FORMULA C24H26O13 | + | FORMULA C24H26O13 |
| − | EXACTMASS 522.137340918 | + | EXACTMASS 522.137340918 |
| − | AVERAGEMASS 522.45544 | + | AVERAGEMASS 522.45544 |
| − | SMILES C(C([C@H](O)1)O[C@@H](Oc(c4O)c(OC)cc(c43)OC(=C(OC)C3=O)c(c2)cc(c(c2)O)OC)[C@H]([C@H]1O)O)O | + | SMILES C(C([C@H](O)1)O[C@@H](Oc(c4O)c(OC)cc(c43)OC(=C(OC)C3=O)c(c2)cc(c(c2)O)OC)[C@H]([C@H]1O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
37 40 0 0 0 0 0 0 0 0999 V2000
-0.7853 0.1638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7853 -0.4786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2290 -0.7997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3273 -0.4786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3273 0.1638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2290 0.4850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8836 -0.7997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4399 -0.4786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4399 0.1638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8836 0.4850 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8836 -1.3006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9960 0.4849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5630 0.1575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1300 0.4849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1300 1.1396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5630 1.4669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9960 1.1396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2290 -1.4419 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1300 1.1396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3414 -0.7996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2299 -1.1272 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-2.8030 -1.6908 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.1882 -1.4517 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.5950 -1.4453 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.0261 -1.0141 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6541 -1.2396 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-3.7789 -1.0791 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4686 -1.7232 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8360 -2.0430 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8893 -0.5138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8673 -0.3051 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8464 2.0430 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2878 2.9403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3059 -0.9786 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1719 -1.4787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1425 0.7826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6424 1.6487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
2 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 20 1 0 0 0 0
26 30 1 0 0 0 0
30 31 1 0 0 0 0
16 32 1 0 0 0 0
32 33 1 0 0 0 0
8 34 1 0 0 0 0
34 35 1 0 0 0 0
1 36 1 0 0 0 0
36 37 1 0 0 0 0
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 30 31
M SBL 4 1 33
M SMT 4 CH2OH
M SVB 4 33 -2.8893 -0.5138
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 36 37
M SBL 3 1 39
M SMT 3 OCH3
M SVB 3 39 -1.1425 0.7826
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 34 35
M SBL 2 1 37
M SMT 2 OCH3
M SVB 2 37 1.6388 -0.6848
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 32 33
M SBL 1 1 35
M SMT 1 OCH3
M SVB 1 35 2.8464 2.043
S SKP 8
ID FL5FECGS0030
KNApSAcK_ID C00005676
NAME Chrysosplenoside C
CAS_RN 24274-45-1
FORMULA C24H26O13
EXACTMASS 522.137340918
AVERAGEMASS 522.45544
SMILES C(C([C@H](O)1)O[C@@H](Oc(c4O)c(OC)cc(c43)OC(=C(OC)C3=O)c(c2)cc(c(c2)O)OC)[C@H]([C@H]1O)O)O
M END