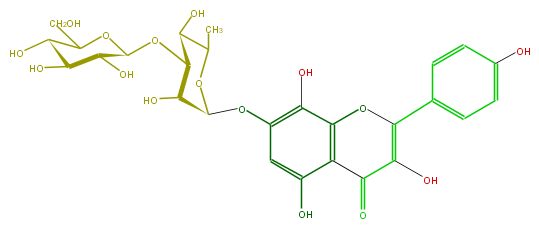
Mol:FL5FFAGS0011
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.0217 -0.2690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0217 -0.2690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0217 -1.0940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0217 -1.0940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7362 -1.5065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7362 -1.5065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4508 -1.0940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4508 -1.0940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4508 -0.2690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4508 -0.2690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7362 0.1436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7362 0.1436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1653 -1.5065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1653 -1.5065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8798 -1.0940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8798 -1.0940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8798 -0.2690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8798 -0.2690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1653 0.1436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1653 0.1436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1653 -2.3501 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1653 -2.3501 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7796 0.3025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7796 0.3025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5077 -0.1181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5077 -0.1181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2360 0.3025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2360 0.3025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2360 1.1433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2360 1.1433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5077 1.5638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5077 1.5638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7796 1.1433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7796 1.1433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7362 -2.3313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7362 -2.3313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8025 1.4703 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8025 1.4703 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6341 -1.5295 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6341 -1.5295 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6419 0.1249 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6419 0.1249 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7362 0.9685 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7362 0.9685 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1024 0.3566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1024 0.3566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4278 -0.0328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4278 -0.0328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6419 0.7162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6419 0.7162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4278 1.4697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4278 1.4697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1024 1.8593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1024 1.8593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8884 1.1102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8884 1.1102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7532 2.3501 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7532 2.3501 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4264 1.9750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4264 1.9750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6588 1.7046 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6588 1.7046 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6915 0.3263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6915 0.3263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1439 1.7030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1439 1.7030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6671 1.0736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6671 1.0736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9806 1.3406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9806 1.3406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3182 1.3477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3182 1.3477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7996 1.8292 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7996 1.8292 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4002 1.5122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4002 1.5122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8025 1.4030 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8025 1.4030 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3472 1.0566 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3472 1.0566 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4003 0.9263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4003 0.9263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0550 2.1056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0550 2.1056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0314 1.8314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0314 1.8314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 21 1 0 0 0 0 | + | 1 21 1 0 0 0 0 |
| − | 6 22 1 0 0 0 0 | + | 6 22 1 0 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 1 0 0 0 | + | 27 28 1 1 0 0 0 |
| − | 28 23 1 1 0 0 0 | + | 28 23 1 1 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 28 31 1 0 0 0 0 | + | 28 31 1 0 0 0 0 |
| − | 23 32 1 0 0 0 0 | + | 23 32 1 0 0 0 0 |
| − | 24 21 1 0 0 0 0 | + | 24 21 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 35 41 1 0 0 0 0 | + | 35 41 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 38 42 1 0 0 0 0 | + | 38 42 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 42 43 | + | M SAL 1 2 42 43 |
| − | M SBL 1 1 47 | + | M SBL 1 1 47 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 47 0.6547 -0.5934 | + | M SBV 1 47 0.6547 -0.5934 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FFAGS0011 | + | ID FL5FFAGS0011 |
| − | FORMULA C27H30O16 | + | FORMULA C27H30O16 |
| − | EXACTMASS 610.153384912 | + | EXACTMASS 610.153384912 |
| − | AVERAGEMASS 610.5175 | + | AVERAGEMASS 610.5175 |
| − | SMILES C(C4O)(C)OC(C(C4OC(O5)C(O)C(O)C(O)C5CO)O)Oc(c3)c(O)c(c1c3O)OC(c(c2)ccc(O)c2)=C(C1=O)O | + | SMILES C(C4O)(C)OC(C(C4OC(O5)C(O)C(O)C(O)C5CO)O)Oc(c3)c(O)c(c1c3O)OC(c(c2)ccc(O)c2)=C(C1=O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
0.0217 -0.2690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0217 -1.0940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7362 -1.5065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4508 -1.0940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4508 -0.2690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7362 0.1436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1653 -1.5065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8798 -1.0940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8798 -0.2690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1653 0.1436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1653 -2.3501 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7796 0.3025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5077 -0.1181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2360 0.3025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2360 1.1433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5077 1.5638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7796 1.1433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7362 -2.3313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.8025 1.4703 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6341 -1.5295 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6419 0.1249 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7362 0.9685 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1024 0.3566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4278 -0.0328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6419 0.7162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4278 1.4697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1024 1.8593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8884 1.1102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7532 2.3501 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4264 1.9750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6588 1.7046 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6915 0.3263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.1439 1.7030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6671 1.0736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9806 1.3406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3182 1.3477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7996 1.8292 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4002 1.5122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.8025 1.4030 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.3472 1.0566 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4003 0.9263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0550 2.1056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0314 1.8314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
20 8 1 0 0 0 0
4 3 1 0 0 0 0
1 21 1 0 0 0 0
6 22 1 0 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 28 1 1 0 0 0
28 23 1 1 0 0 0
27 29 1 0 0 0 0
26 30 1 0 0 0 0
28 31 1 0 0 0 0
23 32 1 0 0 0 0
24 21 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
33 39 1 0 0 0 0
34 40 1 0 0 0 0
35 41 1 0 0 0 0
36 31 1 0 0 0 0
42 43 1 0 0 0 0
38 42 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 42 43
M SBL 1 1 47
M SMT 1 ^CH2OH
M SBV 1 47 0.6547 -0.5934
S SKP 5
ID FL5FFAGS0011
FORMULA C27H30O16
EXACTMASS 610.153384912
AVERAGEMASS 610.5175
SMILES C(C4O)(C)OC(C(C4OC(O5)C(O)C(O)C(O)C5CO)O)Oc(c3)c(O)c(c1c3O)OC(c(c2)ccc(O)c2)=C(C1=O)O
M END