Mol:FL5FGANI0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 32 34 0 0 0 0 0 0 0 0999 V2000 | + | 32 34 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.6850 -0.3209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6850 -0.3209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6850 -0.9633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6850 -0.9633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1287 -1.2845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1287 -1.2845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5724 -0.9633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5724 -0.9633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5724 -0.3209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5724 -0.3209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1287 0.0002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1287 0.0002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0161 -1.2845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0161 -1.2845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5402 -0.9633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5402 -0.9633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5402 -0.3209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5402 -0.3209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0161 0.0002 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0161 0.0002 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0161 -1.7853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0161 -1.7853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0963 0.0001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0963 0.0001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6632 -0.3272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6632 -0.3272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2302 0.0001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2302 0.0001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2302 0.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2302 0.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6632 0.9821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6632 0.9821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0963 0.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0963 0.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1287 -1.9266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1287 -1.9266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7970 0.9821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7970 0.9821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1287 0.6424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1287 0.6424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6848 0.9634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6848 0.9634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6848 1.6055 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -1.6848 1.6055 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -1.1029 1.2346 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1029 1.2346 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2409 1.9266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2409 1.9266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1287 1.9266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1287 1.9266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7970 1.6055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7970 1.6055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4062 -1.4633 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4062 -1.4633 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2722 -1.9633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2722 -1.9633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3728 -0.7492 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3728 -0.7492 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0872 -1.1619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0872 -1.1619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0423 0.2978 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0423 0.2978 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5424 1.1638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5424 1.1638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 6 20 1 0 0 0 0 | + | 6 20 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 21 23 2 0 0 0 0 | + | 21 23 2 0 0 0 0 |
| − | 22 24 1 0 0 0 0 | + | 22 24 1 0 0 0 0 |
| − | 22 25 1 0 0 0 0 | + | 22 25 1 0 0 0 0 |
| − | 24 26 1 0 0 0 0 | + | 24 26 1 0 0 0 0 |
| − | 8 27 1 0 0 0 0 | + | 8 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 2 29 1 0 0 0 0 | + | 2 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 1 31 1 0 0 0 0 | + | 1 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 31 32 | + | M SAL 3 2 31 32 |
| − | M SBL 3 1 33 | + | M SBL 3 1 33 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 33 -2.0423 0.2978 | + | M SVB 3 33 -2.0423 0.2978 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 29 30 | + | M SAL 2 2 29 30 |
| − | M SBL 2 1 31 | + | M SBL 2 1 31 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 31 -2.3995 -0.8719 | + | M SVB 2 31 -2.3995 -0.8719 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 27 28 | + | M SAL 1 2 27 28 |
| − | M SBL 1 1 29 | + | M SBL 1 1 29 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 29 0.739 -1.1696 | + | M SVB 1 29 0.739 -1.1696 |
| − | S SKP 8 | + | S SKP 8 |
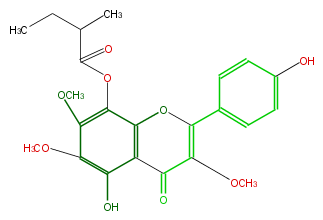
| − | ID FL5FGANI0001 | + | ID FL5FGANI0001 |
| − | KNApSAcK_ID C00004933 | + | KNApSAcK_ID C00004933 |
| − | NAME Pratensin B | + | NAME Pratensin B |
| − | CAS_RN 142609-01-6 | + | CAS_RN 142609-01-6 |
| − | FORMULA C23H24O9 | + | FORMULA C23H24O9 |
| − | EXACTMASS 444.14203236599997 | + | EXACTMASS 444.14203236599997 |
| − | AVERAGEMASS 444.43126 | + | AVERAGEMASS 444.43126 |
| − | SMILES c(c31)(c(O)c(OC)c(c3OC(C(C)CC)=O)OC)C(=O)C(OC)=C(c(c2)ccc(c2)O)O1 | + | SMILES c(c31)(c(O)c(OC)c(c3OC(C(C)CC)=O)OC)C(=O)C(OC)=C(c(c2)ccc(c2)O)O1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
32 34 0 0 0 0 0 0 0 0999 V2000
-1.6850 -0.3209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6850 -0.9633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1287 -1.2845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5724 -0.9633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5724 -0.3209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1287 0.0002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0161 -1.2845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5402 -0.9633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5402 -0.3209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0161 0.0002 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0161 -1.7853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0963 0.0001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6632 -0.3272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2302 0.0001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2302 0.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6632 0.9821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0963 0.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1287 -1.9266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7970 0.9821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1287 0.6424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6848 0.9634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6848 1.6055 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-1.1029 1.2346 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2409 1.9266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1287 1.9266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7970 1.6055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4062 -1.4633 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2722 -1.9633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3728 -0.7492 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0872 -1.1619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0423 0.2978 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5424 1.1638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
15 19 1 0 0 0 0
6 20 1 0 0 0 0
20 21 1 0 0 0 0
21 22 1 0 0 0 0
21 23 2 0 0 0 0
22 24 1 0 0 0 0
22 25 1 0 0 0 0
24 26 1 0 0 0 0
8 27 1 0 0 0 0
27 28 1 0 0 0 0
2 29 1 0 0 0 0
29 30 1 0 0 0 0
1 31 1 0 0 0 0
31 32 1 0 0 0 0
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 31 32
M SBL 3 1 33
M SMT 3 OCH3
M SVB 3 33 -2.0423 0.2978
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 29 30
M SBL 2 1 31
M SMT 2 OCH3
M SVB 2 31 -2.3995 -0.8719
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 27 28
M SBL 1 1 29
M SMT 1 OCH3
M SVB 1 29 0.739 -1.1696
S SKP 8
ID FL5FGANI0001
KNApSAcK_ID C00004933
NAME Pratensin B
CAS_RN 142609-01-6
FORMULA C23H24O9
EXACTMASS 444.14203236599997
AVERAGEMASS 444.43126
SMILES c(c31)(c(O)c(OC)c(c3OC(C(C)CC)=O)OC)C(=O)C(OC)=C(c(c2)ccc(c2)O)O1
M END