Mol:FL5FGGNS0006
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 30 32 0 0 0 0 0 0 0 0999 V2000 | + | 30 32 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.6455 -1.8975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6455 -1.8975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3243 -1.3412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3243 -1.3412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6455 -0.7849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6455 -0.7849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2878 -0.7849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2878 -0.7849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6090 -1.3412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6090 -1.3412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2878 -1.8975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2878 -1.8975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6090 -0.2286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6090 -0.2286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2514 -0.2286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2514 -0.2286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5725 -0.7849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5725 -0.7849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2514 -1.3412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2514 -1.3412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3586 0.2052 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3586 0.2052 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2147 -0.7849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2147 -0.7849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5420 -0.2178 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5420 -0.2178 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.1967 -0.2179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.1967 -0.2179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.5240 -0.7849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.5240 -0.7849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.1967 -1.3518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.1967 -1.3518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5420 -1.3519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5420 -1.3519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3244 -2.4536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3244 -2.4536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3244 -0.2288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3244 -0.2288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.3900 -0.2849 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.3900 -0.2849 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8357 -1.8705 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8357 -1.8705 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1180 -1.0952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1180 -1.0952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7514 0.6374 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7514 0.6374 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2515 1.5034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2515 1.5034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.7302 -1.7090 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.7302 -1.7090 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.5611 -2.2654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.5611 -2.2654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.6966 0.6481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.6966 0.6481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.1966 1.5141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.1966 1.5141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8151 -2.2547 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8151 -2.2547 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6430 -2.8156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6430 -2.8156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 2 21 1 0 0 0 0 | + | 2 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 8 23 1 0 0 0 0 | + | 8 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 16 25 1 0 0 0 0 | + | 16 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 14 27 1 0 0 0 0 | + | 14 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 6 29 1 0 0 0 0 | + | 6 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | M STY 1 5 SUP | + | M STY 1 5 SUP |
| − | M SLB 1 5 5 | + | M SLB 1 5 5 |
| − | M SAL 5 2 29 30 | + | M SAL 5 2 29 30 |
| − | M SBL 5 1 31 | + | M SBL 5 1 31 |
| − | M SMT 5 OCH3 | + | M SMT 5 OCH3 |
| − | M SVB 5 31 -1.2281 0.7574 | + | M SVB 5 31 -1.2281 0.7574 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 27 28 | + | M SAL 4 2 27 28 |
| − | M SBL 4 1 29 | + | M SBL 4 1 29 |
| − | M SMT 4 OCH3 | + | M SMT 4 OCH3 |
| − | M SVB 4 29 2.0624 -0.022 | + | M SVB 4 29 2.0624 -0.022 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 25 26 | + | M SAL 3 2 25 26 |
| − | M SBL 3 1 27 | + | M SBL 3 1 27 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 27 1.5693 1.7424 | + | M SVB 3 27 1.5693 1.7424 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 23 24 | + | M SAL 2 2 23 24 |
| − | M SBL 2 1 25 | + | M SBL 2 1 25 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 25 0.3617 -0.9854 | + | M SVB 2 25 0.3617 -0.9854 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 21 22 | + | M SAL 1 2 21 22 |
| − | M SBL 1 1 23 | + | M SBL 1 1 23 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 23 -2.7769 -0.6877 | + | M SVB 1 23 -2.7769 -0.6877 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FGGNS0006 | + | ID FL5FGGNS0006 |
| − | KNApSAcK_ID C00004863 | + | KNApSAcK_ID C00004863 |
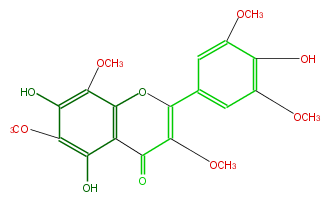
| − | NAME 5,7,4'-Trihydroxy-3,6,8,3',5'-pentamethoxyflavone | + | NAME 5,7,4'-Trihydroxy-3,6,8,3',5'-pentamethoxyflavone |
| − | CAS_RN 96887-19-3 | + | CAS_RN 96887-19-3 |
| − | FORMULA C20H20O10 | + | FORMULA C20H20O10 |
| − | EXACTMASS 420.10564686 | + | EXACTMASS 420.10564686 |
| − | AVERAGEMASS 420.3668 | + | AVERAGEMASS 420.3668 |
| − | SMILES C(O2)(c(c3)cc(OC)c(O)c3OC)=C(C(c(c21)c(c(c(O)c1OC)OC)O)=O)OC | + | SMILES C(O2)(c(c3)cc(OC)c(O)c3OC)=C(C(c(c21)c(c(c(O)c1OC)OC)O)=O)OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/ 30 32 0 0 0 0 0 0 0 0999 V2000 -2.6455 -1.8975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -2.3243 -1.3412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -2.6455 -0.7849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -3.2878 -0.7849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -3.6090 -1.3412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -3.2878 -1.8975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -3.6090 -0.2286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -4.2514 -0.2286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -4.5725 -0.7849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -4.2514 -1.3412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 -3.3586 0.2052 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 -5.2147 -0.7849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -5.5420 -0.2178 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -6.1967 -0.2179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -6.5240 -0.7849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -6.1967 -1.3518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -5.5420 -1.3519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -2.3244 -2.4536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 -2.3244 -0.2288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 -7.3900 -0.2849 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 -1.8357 -1.8705 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 -2.1180 -1.0952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -4.7514 0.6374 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 -5.2515 1.5034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -6.7302 -1.7090 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 -7.5611 -2.2654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -6.6966 0.6481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 -7.1966 1.5141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -3.8151 -2.2547 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 -4.6430 -2.8156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 1 2 1 0 0 0 0 2 3 2 0 0 0 0 3 4 1 0 0 0 0 4 5 2 0 0 0 0 5 6 1 0 0 0 0 6 1 2 0 0 0 0 4 7 1 0 0 0 0 7 8 1 0 0 0 0 8 9 2 0 0 0 0 9 10 1 0 0 0 0 10 5 1 0 0 0 0 7 11 2 0 0 0 0 9 12 1 0 0 0 0 12 13 2 0 0 0 0 13 14 1 0 0 0 0 14 15 2 0 0 0 0 15 16 1 0 0 0 0 16 17 2 0 0 0 0 17 12 1 0 0 0 0 1 18 1 0 0 0 0 3 19 1 0 0 0 0 15 20 1 0 0 0 0 2 21 1 0 0 0 0 21 22 1 0 0 0 0 8 23 1 0 0 0 0 23 24 1 0 0 0 0 16 25 1 0 0 0 0 25 26 1 0 0 0 0 14 27 1 0 0 0 0 27 28 1 0 0 0 0 6 29 1 0 0 0 0 29 30 1 0 0 0 0 M STY 1 5 SUP M SLB 1 5 5 M SAL 5 2 29 30 M SBL 5 1 31 M SMT 5 OCH3 M SVB 5 31 -1.2281 0.7574 M STY 1 4 SUP M SLB 1 4 4 M SAL 4 2 27 28 M SBL 4 1 29 M SMT 4 OCH3 M SVB 4 29 2.0624 -0.022 M STY 1 3 SUP M SLB 1 3 3 M SAL 3 2 25 26 M SBL 3 1 27 M SMT 3 OCH3 M SVB 3 27 1.5693 1.7424 M STY 1 2 SUP M SLB 1 2 2 M SAL 2 2 23 24 M SBL 2 1 25 M SMT 2 OCH3 M SVB 2 25 0.3617 -0.9854 M STY 1 1 SUP M SLB 1 1 1 M SAL 1 2 21 22 M SBL 1 1 23 M SMT 1 OCH3 M SVB 1 23 -2.7769 -0.6877 S SKP 8 ID FL5FGGNS0006 KNApSAcK_ID C00004863 NAME 5,7,4'-Trihydroxy-3,6,8,3',5'-pentamethoxyflavone CAS_RN 96887-19-3 FORMULA C20H20O10 EXACTMASS 420.10564686 AVERAGEMASS 420.3668 SMILES C(O2)(c(c3)cc(OC)c(O)c3OC)=C(C(c(c21)c(c(c(O)c1OC)OC)O)=O)OC M END