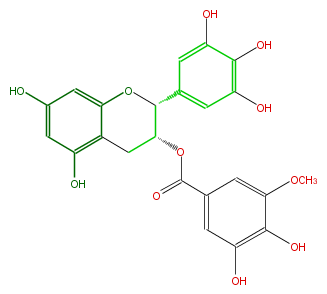
Mol:FL63AGNS0005
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 34 37 0 0 0 0 0 0 0 0999 V2000 | + | 34 37 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.5545 0.8219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5545 0.8219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5545 0.2001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5545 0.2001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0160 -0.1108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0160 -0.1108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4774 0.2001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4774 0.2001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4774 0.8219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4774 0.8219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0160 1.1328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0160 1.1328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9389 -0.1108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9389 -0.1108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4004 0.2001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4004 0.2001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4004 0.8219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4004 0.8219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9389 1.1328 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9389 1.1328 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0927 1.1327 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0927 1.1327 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1106 -0.0949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1106 -0.0949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0792 1.0988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0792 1.0988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6239 0.7843 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6239 0.7843 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1686 1.0988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1686 1.0988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1686 1.7278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1686 1.7278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6239 2.0423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6239 2.0423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0792 1.7278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0792 1.7278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0160 -0.7323 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0160 -0.7323 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7128 2.0420 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7128 2.0420 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6239 2.6706 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6239 2.6706 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7128 0.7846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7128 0.7846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1106 -0.6848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1106 -0.6848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3717 -0.9633 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3717 -0.9633 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6203 -0.9790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6203 -0.9790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6203 -1.6558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6203 -1.6558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2063 -1.9941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2063 -1.9941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7924 -1.6558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7924 -1.6558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7924 -0.9790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7924 -0.9790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2063 -0.6407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2063 -0.6407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2063 -2.6706 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2063 -2.6706 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3782 -1.9940 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3782 -1.9940 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3782 -0.6408 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3782 -0.6408 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0927 -1.0533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0927 -1.0533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 1 11 1 0 0 0 0 | + | 1 11 1 0 0 0 0 |
| − | 8 12 1 6 0 0 0 | + | 8 12 1 6 0 0 0 |
| − | 9 13 1 6 0 0 0 | + | 9 13 1 6 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 17 21 1 0 0 0 0 | + | 17 21 1 0 0 0 0 |
| − | 15 22 1 0 0 0 0 | + | 15 22 1 0 0 0 0 |
| − | 12 23 1 0 0 0 0 | + | 12 23 1 0 0 0 0 |
| − | 23 24 2 0 0 0 0 | + | 23 24 2 0 0 0 0 |
| − | 23 25 1 0 0 0 0 | + | 23 25 1 0 0 0 0 |
| − | 25 26 2 0 0 0 0 | + | 25 26 2 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 2 0 0 0 0 | + | 27 28 2 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 2 0 0 0 0 | + | 29 30 2 0 0 0 0 |
| − | 30 25 1 0 0 0 0 | + | 30 25 1 0 0 0 0 |
| − | 27 31 1 0 0 0 0 | + | 27 31 1 0 0 0 0 |
| − | 28 32 1 0 0 0 0 | + | 28 32 1 0 0 0 0 |
| − | 29 33 1 0 0 0 0 | + | 29 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 33 34 | + | M SAL 1 2 33 34 |
| − | M SBL 1 1 36 | + | M SBL 1 1 36 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 36 -6.7289 5.1202 | + | M SBV 1 36 -6.7289 5.1202 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL63AGNS0005 | + | ID FL63AGNS0005 |
| − | KNApSAcK_ID C00008883 | + | KNApSAcK_ID C00008883 |
| − | NAME Epigallocatechin 3-O-(3-O-methylgallate) | + | NAME Epigallocatechin 3-O-(3-O-methylgallate) |
| − | CAS_RN 83104-87-4 | + | CAS_RN 83104-87-4 |
| − | FORMULA C23H20O11 | + | FORMULA C23H20O11 |
| − | EXACTMASS 472.100561482 | + | EXACTMASS 472.100561482 |
| − | AVERAGEMASS 472.3983 | + | AVERAGEMASS 472.3983 |
| − | SMILES c(c1)(O)cc(O3)c(CC(C(c(c4)cc(O)c(O)c(O)4)3)OC(c(c2)cc(OC)c(c(O)2)O)=O)c(O)1 | + | SMILES c(c1)(O)cc(O3)c(CC(C(c(c4)cc(O)c(O)c(O)4)3)OC(c(c2)cc(OC)c(c(O)2)O)=O)c(O)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
34 37 0 0 0 0 0 0 0 0999 V2000
-2.5545 0.8219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5545 0.2001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0160 -0.1108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4774 0.2001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4774 0.8219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0160 1.1328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9389 -0.1108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4004 0.2001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4004 0.8219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9389 1.1328 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0927 1.1327 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1106 -0.0949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0792 1.0988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6239 0.7843 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1686 1.0988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1686 1.7278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6239 2.0423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0792 1.7278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0160 -0.7323 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7128 2.0420 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6239 2.6706 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7128 0.7846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1106 -0.6848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3717 -0.9633 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6203 -0.9790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6203 -1.6558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2063 -1.9941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7924 -1.6558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7924 -0.9790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2063 -0.6407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2063 -2.6706 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3782 -1.9940 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3782 -0.6408 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0927 -1.0533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
1 11 1 0 0 0 0
8 12 1 6 0 0 0
9 13 1 6 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
3 19 1 0 0 0 0
16 20 1 0 0 0 0
17 21 1 0 0 0 0
15 22 1 0 0 0 0
12 23 1 0 0 0 0
23 24 2 0 0 0 0
23 25 1 0 0 0 0
25 26 2 0 0 0 0
26 27 1 0 0 0 0
27 28 2 0 0 0 0
28 29 1 0 0 0 0
29 30 2 0 0 0 0
30 25 1 0 0 0 0
27 31 1 0 0 0 0
28 32 1 0 0 0 0
29 33 1 0 0 0 0
33 34 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 33 34
M SBL 1 1 36
M SMT 1 OCH3
M SBV 1 36 -6.7289 5.1202
S SKP 8
ID FL63AGNS0005
KNApSAcK_ID C00008883
NAME Epigallocatechin 3-O-(3-O-methylgallate)
CAS_RN 83104-87-4
FORMULA C23H20O11
EXACTMASS 472.100561482
AVERAGEMASS 472.3983
SMILES c(c1)(O)cc(O3)c(CC(C(c(c4)cc(O)c(O)c(O)4)3)OC(c(c2)cc(OC)c(c(O)2)O)=O)c(O)1
M END