Mol:FL7AAAGL0005
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -4.4786 0.4576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4786 0.4576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4786 -0.1848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4786 -0.1848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9223 -0.5059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9223 -0.5059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3660 -0.1848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3660 -0.1848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3660 0.4576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3660 0.4576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9223 0.7788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9223 0.7788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8097 -0.5059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8097 -0.5059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2534 -0.1848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2534 -0.1848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2534 0.4576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2534 0.4576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8097 0.7788 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -2.8097 0.7788 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6973 0.7787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6973 0.7787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1303 0.4513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1303 0.4513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5633 0.7787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5633 0.7787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5633 1.4334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5633 1.4334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1303 1.7607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1303 1.7607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6973 1.4334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6973 1.4334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0035 1.7606 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0035 1.7606 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0347 0.7787 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0347 0.7787 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9223 -1.1481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9223 -1.1481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6006 -0.5209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6006 -0.5209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5186 -0.2024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5186 -0.2024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9062 -0.8738 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9062 -0.8738 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1608 -0.6608 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1608 -0.6608 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5892 -0.8738 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5892 -0.8738 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9769 -0.2024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9769 -0.2024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2314 -0.4154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2314 -0.4154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1848 -0.2024 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1848 -0.2024 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5384 0.1163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5384 0.1163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4398 -0.3264 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4398 -0.3264 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3000 -0.8738 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3000 -0.8738 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6994 -1.4077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6994 -1.4077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7814 -1.0893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7814 -1.0893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3938 -1.7607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3938 -1.7607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1392 -1.5476 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1392 -1.5476 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8892 -1.7607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8892 -1.7607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2769 -1.0893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2769 -1.0893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5314 -1.3023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5314 -1.3023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1152 -1.0893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1152 -1.0893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8384 -0.7706 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8384 -0.7706 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7454 -1.0893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7454 -1.0893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6000 -1.7607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6000 -1.7607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0347 -1.7607 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0347 -1.7607 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 22 20 1 0 0 0 0 | + | 22 20 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 37 39 1 0 0 0 0 | + | 37 39 1 0 0 0 0 |
| − | 36 40 1 0 0 0 0 | + | 36 40 1 0 0 0 0 |
| − | 35 41 1 0 0 0 0 | + | 35 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 33 31 1 0 0 0 0 | + | 33 31 1 0 0 0 0 |
| − | 31 30 1 0 0 0 0 | + | 31 30 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AAAGL0005 | + | ID FL7AAAGL0005 |
| − | KNApSAcK_ID C00006637 | + | KNApSAcK_ID C00006637 |
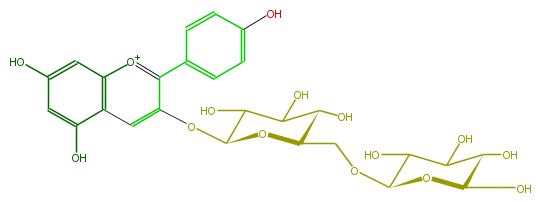
| − | NAME Pelargonidin 3-gentiobioside | + | NAME Pelargonidin 3-gentiobioside |
| − | CAS_RN 103102-91-6 | + | CAS_RN 103102-91-6 |
| − | FORMULA C27H31O15 | + | FORMULA C27H31O15 |
| − | EXACTMASS 595.166295322 | + | EXACTMASS 595.166295322 |
| − | AVERAGEMASS 595.52604 | + | AVERAGEMASS 595.52604 |
| − | SMILES c(O)(c5)ccc(c5)c([o+1]2)c(OC(O3)C(O)C(O)C(O)C3COC(C4O)OC(C(C(O)4)O)CO)cc(c12)c(cc(c1)O)O | + | SMILES c(O)(c5)ccc(c5)c([o+1]2)c(OC(O3)C(O)C(O)C(O)C3COC(C4O)OC(C(C(O)4)O)CO)cc(c12)c(cc(c1)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-4.4786 0.4576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4786 -0.1848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9223 -0.5059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3660 -0.1848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3660 0.4576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9223 0.7788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8097 -0.5059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2534 -0.1848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2534 0.4576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8097 0.7788 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
-1.6973 0.7787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1303 0.4513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5633 0.7787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5633 1.4334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1303 1.7607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6973 1.4334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0035 1.7606 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0347 0.7787 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9223 -1.1481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6006 -0.5209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5186 -0.2024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9062 -0.8738 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1608 -0.6608 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5892 -0.8738 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9769 -0.2024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2314 -0.4154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1848 -0.2024 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5384 0.1163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4398 -0.3264 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3000 -0.8738 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6994 -1.4077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7814 -1.0893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3938 -1.7607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1392 -1.5476 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8892 -1.7607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2769 -1.0893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5314 -1.3023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1152 -1.0893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8384 -0.7706 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7454 -1.0893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6000 -1.7607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0347 -1.7607 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
14 17 1 0 0 0 0
1 18 1 0 0 0 0
3 19 1 0 0 0 0
8 20 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
24 30 1 0 0 0 0
22 20 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
32 38 1 0 0 0 0
37 39 1 0 0 0 0
36 40 1 0 0 0 0
35 41 1 0 0 0 0
41 42 1 0 0 0 0
33 31 1 0 0 0 0
31 30 1 0 0 0 0
S SKP 8
ID FL7AAAGL0005
KNApSAcK_ID C00006637
NAME Pelargonidin 3-gentiobioside
CAS_RN 103102-91-6
FORMULA C27H31O15
EXACTMASS 595.166295322
AVERAGEMASS 595.52604
SMILES c(O)(c5)ccc(c5)c([o+1]2)c(OC(O3)C(O)C(O)C(O)C3COC(C4O)OC(C(C(O)4)O)CO)cc(c12)c(cc(c1)O)O
M END