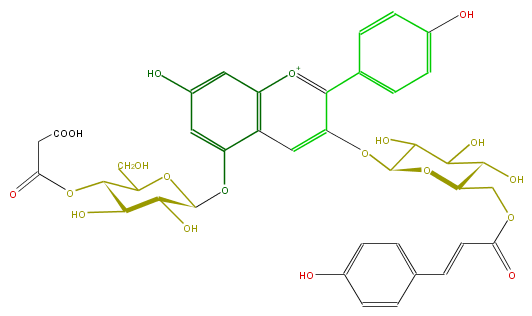
Mol:FL7AAAGL0034
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 59 64 0 0 0 0 0 0 0 0999 V2000 | + | 59 64 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.6141 1.5011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6141 1.5011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6141 0.6251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6141 0.6251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8555 0.1871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8555 0.1871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0969 0.6251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0969 0.6251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0969 1.5011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0969 1.5011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8555 1.9390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8555 1.9390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6616 0.1871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6616 0.1871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4202 0.6251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4202 0.6251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4202 1.5011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4202 1.5011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6616 1.9390 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 0.6616 1.9390 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1785 1.9388 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1785 1.9388 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9515 1.4925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9515 1.4925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7247 1.9388 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7247 1.9388 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7247 2.8316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7247 2.8316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9515 3.2779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9515 3.2779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1785 2.8316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1785 2.8316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3724 1.9388 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3724 1.9388 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4976 3.2779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4976 3.2779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8555 -0.6884 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8555 -0.6884 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2997 0.1553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2997 0.1553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4382 0.3215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4382 0.3215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8581 -0.2587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8581 -0.2587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6784 -0.2450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6784 -0.2450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3865 -0.6683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3865 -0.6683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9668 -0.0881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9668 -0.0881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1465 -0.1017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1465 -0.1017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7554 0.4425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7554 0.4425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7414 0.3835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7414 0.3835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6219 -0.4490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6219 -0.4490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1577 -0.6531 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1577 -0.6531 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5911 -0.5092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5911 -0.5092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0849 -1.1773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0849 -1.1773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3561 -0.8939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3561 -0.8939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5229 -1.0957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5229 -1.0957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1639 -0.3752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1639 -0.3752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8016 -0.7117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8016 -0.7117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3336 -0.7493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3336 -0.7493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0711 -1.2168 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0711 -1.2168 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7103 -1.5344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7103 -1.5344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5658 -1.3001 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5658 -1.3001 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1903 -1.8997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1903 -1.8997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4879 -1.8997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4879 -1.8997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0864 -2.5948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0864 -2.5948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4192 -2.5948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4192 -2.5948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0249 -1.9118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0249 -1.9118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2362 -1.9118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2362 -1.9118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8418 -2.5948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8418 -2.5948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2362 -3.2779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2362 -3.2779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0249 -3.2779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0249 -3.2779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0544 -2.5948 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0544 -2.5948 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5935 -2.5980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5935 -2.5980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0474 -0.3232 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0474 -0.3232 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6219 -0.7697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6219 -0.7697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0425 0.3786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0425 0.3786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3252 -0.1353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3252 -0.1353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6139 -0.5810 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6139 -0.5810 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6483 0.5902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6483 0.5902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9953 1.3344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9953 1.3344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1195 -0.0825 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1195 -0.0825 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 22 20 1 0 0 0 0 | + | 22 20 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 19 1 0 0 0 0 | + | 34 19 1 0 0 0 0 |
| − | 30 40 1 0 0 0 0 | + | 30 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 42 43 2 0 0 0 0 | + | 42 43 2 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 2 0 0 0 0 | + | 44 45 2 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 2 0 0 0 0 | + | 46 47 2 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 2 0 0 0 0 | + | 48 49 2 0 0 0 0 |
| − | 49 44 1 0 0 0 0 | + | 49 44 1 0 0 0 0 |
| − | 47 50 1 0 0 0 0 | + | 47 50 1 0 0 0 0 |
| − | 41 51 2 0 0 0 0 | + | 41 51 2 0 0 0 0 |
| − | 37 52 1 0 0 0 0 | + | 37 52 1 0 0 0 0 |
| − | 52 53 2 0 0 0 0 | + | 52 53 2 0 0 0 0 |
| − | 52 54 1 0 0 0 0 | + | 52 54 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 36 55 1 0 0 0 0 | + | 36 55 1 0 0 0 0 |
| − | 57 58 2 0 0 0 0 | + | 57 58 2 0 0 0 0 |
| − | 57 59 1 0 0 0 0 | + | 57 59 1 0 0 0 0 |
| − | 54 57 1 0 0 0 0 | + | 54 57 1 0 0 0 0 |
| − | M CHG 1 10 1 | + | M CHG 1 10 1 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 55 56 | + | M SAL 1 2 55 56 |
| − | M SBL 1 1 61 | + | M SBL 1 1 61 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 61 0.5236 -0.5765 | + | M SBV 1 61 0.5236 -0.5765 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 3 57 58 59 | + | M SAL 2 3 57 58 59 |
| − | M SBL 2 1 64 | + | M SBL 2 1 64 |
| − | M SMT 2 COOH | + | M SMT 2 COOH |
| − | M SBV 2 64 -0.3942 -0.2117 | + | M SBV 2 64 -0.3942 -0.2117 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL7AAAGL0034 | + | ID FL7AAAGL0034 |
| − | FORMULA C39H39O20 | + | FORMULA C39H39O20 |
| − | EXACTMASS 827.2034686879999 | + | EXACTMASS 827.2034686879999 |
| − | AVERAGEMASS 827.71496 | + | AVERAGEMASS 827.71496 |
| − | SMILES C(O)(=O)CC(OC(C(CO)1)C(C(C(Oc(c2)c(c3)c([o+1]c(c(c6)ccc(c6)O)c3OC(O4)C(C(C(O)C4COC(C=Cc(c5)ccc(O)c5)=O)O)O)cc2O)O1)O)O)=O | + | SMILES C(O)(=O)CC(OC(C(CO)1)C(C(C(Oc(c2)c(c3)c([o+1]c(c(c6)ccc(c6)O)c3OC(O4)C(C(C(O)C4COC(C=Cc(c5)ccc(O)c5)=O)O)O)cc2O)O1)O)O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
59 64 0 0 0 0 0 0 0 0999 V2000
-1.6141 1.5011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6141 0.6251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8555 0.1871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0969 0.6251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0969 1.5011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8555 1.9390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6616 0.1871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4202 0.6251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4202 1.5011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6616 1.9390 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
2.1785 1.9388 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9515 1.4925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7247 1.9388 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7247 2.8316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9515 3.2779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1785 2.8316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3724 1.9388 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4976 3.2779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8555 -0.6884 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2997 0.1553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4382 0.3215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8581 -0.2587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6784 -0.2450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3865 -0.6683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9668 -0.0881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1465 -0.1017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7554 0.4425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7414 0.3835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.6219 -0.4490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.1577 -0.6531 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5911 -0.5092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0849 -1.1773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3561 -0.8939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5229 -1.0957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1639 -0.3752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8016 -0.7117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3336 -0.7493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0711 -1.2168 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7103 -1.5344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.5658 -1.3001 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.1903 -1.8997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4879 -1.8997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0864 -2.5948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4192 -2.5948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0249 -1.9118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2362 -1.9118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8418 -2.5948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2362 -3.2779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0249 -3.2779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0544 -2.5948 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.5935 -2.5980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0474 -0.3232 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.6219 -0.7697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0425 0.3786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3252 -0.1353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6139 -0.5810 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6483 0.5902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9953 1.3344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.1195 -0.0825 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
24 30 1 0 0 0 0
22 20 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 19 1 0 0 0 0
30 40 1 0 0 0 0
40 41 1 0 0 0 0
41 42 1 0 0 0 0
42 43 2 0 0 0 0
43 44 1 0 0 0 0
44 45 2 0 0 0 0
45 46 1 0 0 0 0
46 47 2 0 0 0 0
47 48 1 0 0 0 0
48 49 2 0 0 0 0
49 44 1 0 0 0 0
47 50 1 0 0 0 0
41 51 2 0 0 0 0
37 52 1 0 0 0 0
52 53 2 0 0 0 0
52 54 1 0 0 0 0
55 56 1 0 0 0 0
36 55 1 0 0 0 0
57 58 2 0 0 0 0
57 59 1 0 0 0 0
54 57 1 0 0 0 0
M CHG 1 10 1
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 55 56
M SBL 1 1 61
M SMT 1 ^ CH2OH
M SBV 1 61 0.5236 -0.5765
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 3 57 58 59
M SBL 2 1 64
M SMT 2 COOH
M SBV 2 64 -0.3942 -0.2117
S SKP 5
ID FL7AAAGL0034
FORMULA C39H39O20
EXACTMASS 827.2034686879999
AVERAGEMASS 827.71496
SMILES C(O)(=O)CC(OC(C(CO)1)C(C(C(Oc(c2)c(c3)c([o+1]c(c(c6)ccc(c6)O)c3OC(O4)C(C(C(O)C4COC(C=Cc(c5)ccc(O)c5)=O)O)O)cc2O)O1)O)O)=O
M END