Mol:FL7AAAGL0062
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 66 72 0 0 0 0 0 0 0 0999 V2000 | + | 66 72 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.2201 2.1814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2201 2.1814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2201 1.3564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2201 1.3564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5056 0.9439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5056 0.9439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2089 1.3564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2089 1.3564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2089 2.1814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2089 2.1814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5056 2.5939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5056 2.5939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9233 0.9439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9233 0.9439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6378 1.3564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6378 1.3564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6378 2.1814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6378 2.1814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9233 2.5939 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 0.9233 2.5939 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4143 2.6297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4143 2.6297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1288 2.2172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1288 2.2172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8433 2.6297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8433 2.6297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8433 3.4547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8433 3.4547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1288 3.8672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1288 3.8672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4143 3.4547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4143 3.4547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4835 3.8243 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4835 3.8243 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8001 2.5162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8001 2.5162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5056 0.1812 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5056 0.1812 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2880 0.9810 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2880 0.9810 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9681 0.2134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9681 0.2134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5555 -0.5013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5555 -0.5013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7572 -0.2922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7572 -0.2922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9637 -0.5190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9637 -0.5190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3763 0.1957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3763 0.1957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1747 -0.0133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1747 -0.0133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3316 0.5725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3316 0.5725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8115 0.8495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8115 0.8495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4513 -0.2698 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4513 -0.2698 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0042 -0.2423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0042 -0.2423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9286 -0.9321 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9286 -0.9321 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1118 -1.1141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1118 -1.1141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9369 -1.1312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9369 -1.1312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1713 -0.3399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1713 -0.3399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7763 0.2215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7763 0.2215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9512 0.2386 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9512 0.2386 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7166 -0.5527 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7166 -0.5527 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1342 -0.3836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1342 -0.3836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6487 -0.6507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6487 -0.6507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2750 -1.7777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2750 -1.7777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4829 -1.3809 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4829 -1.3809 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6300 -0.8180 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6300 -0.8180 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3417 -1.5580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3417 -1.5580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7673 -2.2953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7673 -2.2953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4822 -1.5580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4822 -1.5580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8941 -2.2715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8941 -2.2715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7180 -2.2715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7180 -2.2715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1305 -1.5571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1305 -1.5571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9555 -1.5571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9555 -1.5571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3680 -2.2715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3680 -2.2715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9555 -2.9860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9555 -2.9860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1305 -2.9860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1305 -2.9860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1919 -2.2715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1919 -2.2715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9317 -3.2637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9317 -3.2637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7359 -3.0785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7359 -3.0785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7697 -2.2539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7697 -2.2539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2191 -1.5617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2191 -1.5617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4148 -1.7468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4148 -1.7468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3809 -2.5714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3809 -2.5714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7747 -2.5499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7747 -2.5499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3693 -2.9275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3693 -2.9275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2521 -3.8672 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2521 -3.8672 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3568 -3.4316 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3568 -3.4316 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3314 -2.6053 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3314 -2.6053 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5356 -3.5661 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5356 -3.5661 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4835 -3.5661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4835 -3.5661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 21 29 1 0 0 0 0 | + | 21 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 32 40 1 0 0 0 0 | + | 32 40 1 0 0 0 0 |
| − | 33 41 1 0 0 0 0 | + | 33 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 35 20 1 0 0 0 0 | + | 35 20 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 43 45 1 0 0 0 0 | + | 43 45 1 0 0 0 0 |
| − | 45 46 2 0 0 0 0 | + | 45 46 2 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 2 0 0 0 0 | + | 47 48 2 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 51 52 2 0 0 0 0 | + | 51 52 2 0 0 0 0 |
| − | 52 47 1 0 0 0 0 | + | 52 47 1 0 0 0 0 |
| − | 50 53 1 0 0 0 0 | + | 50 53 1 0 0 0 0 |
| − | 54 55 1 1 0 0 0 | + | 54 55 1 1 0 0 0 |
| − | 55 56 1 1 0 0 0 | + | 55 56 1 1 0 0 0 |
| − | 57 56 1 1 0 0 0 | + | 57 56 1 1 0 0 0 |
| − | 57 58 1 0 0 0 0 | + | 57 58 1 0 0 0 0 |
| − | 58 59 1 0 0 0 0 | + | 58 59 1 0 0 0 0 |
| − | 59 54 1 0 0 0 0 | + | 59 54 1 0 0 0 0 |
| − | 59 60 1 0 0 0 0 | + | 59 60 1 0 0 0 0 |
| − | 60 61 1 0 0 0 0 | + | 60 61 1 0 0 0 0 |
| − | 54 62 1 0 0 0 0 | + | 54 62 1 0 0 0 0 |
| − | 55 63 1 0 0 0 0 | + | 55 63 1 0 0 0 0 |
| − | 56 64 1 0 0 0 0 | + | 56 64 1 0 0 0 0 |
| − | 57 42 1 0 0 0 0 | + | 57 42 1 0 0 0 0 |
| − | 43 39 1 0 0 0 0 | + | 43 39 1 0 0 0 0 |
| − | 51 65 1 0 0 0 0 | + | 51 65 1 0 0 0 0 |
| − | 65 66 1 0 0 0 0 | + | 65 66 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AAAGL0062 | + | ID FL7AAAGL0062 |
| − | KNApSAcK_ID C00014838 | + | KNApSAcK_ID C00014838 |
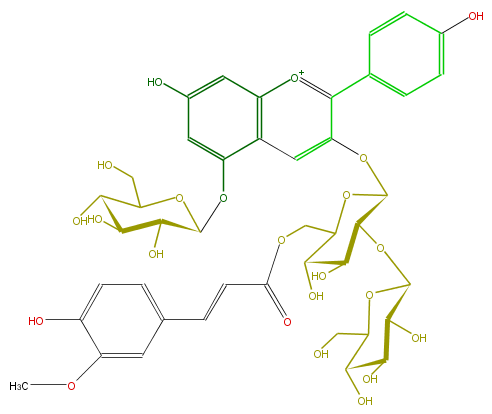
| − | NAME Pelargonidin 3-O-[6-O-(E)-feruloyl-2-O-beta-D-glucopyranosyl-beta-D-glucopyranoside]-5-O-(beta-D-glucopyranoside);Pelargonidin 3-(6-ferulyl-2-glucosylglucoside)-5-glucoside | + | NAME Pelargonidin 3-O-[6-O-(E)-feruloyl-2-O-beta-D-glucopyranosyl-beta-D-glucopyranoside]-5-O-(beta-D-glucopyranoside);Pelargonidin 3-(6-ferulyl-2-glucosylglucoside)-5-glucoside |
| − | CAS_RN 185027-87-6 | + | CAS_RN 185027-87-6 |
| − | FORMULA C43H49O23 | + | FORMULA C43H49O23 |
| − | EXACTMASS 933.266462874 | + | EXACTMASS 933.266462874 |
| − | AVERAGEMASS 933.83536 | + | AVERAGEMASS 933.83536 |
| − | SMILES c(O)(c2)cc(c(c4)c([o+1]c(c4OC(C5OC(C(O)7)OC(C(O)C7O)CO)OC(COC(=O)C=Cc(c6)cc(OC)c(c6)O)C(C(O)5)O)c(c3)ccc(O)c3)2)OC(C1O)OC(CO)C(C1O)O | + | SMILES c(O)(c2)cc(c(c4)c([o+1]c(c4OC(C5OC(C(O)7)OC(C(O)C7O)CO)OC(COC(=O)C=Cc(c6)cc(OC)c(c6)O)C(C(O)5)O)c(c3)ccc(O)c3)2)OC(C1O)OC(CO)C(C1O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
66 72 0 0 0 0 0 0 0 0999 V2000
-1.2201 2.1814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2201 1.3564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5056 0.9439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2089 1.3564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2089 2.1814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5056 2.5939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9233 0.9439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6378 1.3564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6378 2.1814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9233 2.5939 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
2.4143 2.6297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1288 2.2172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8433 2.6297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8433 3.4547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1288 3.8672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4143 3.4547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4835 3.8243 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8001 2.5162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5056 0.1812 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2880 0.9810 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9681 0.2134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5555 -0.5013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7572 -0.2922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9637 -0.5190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3763 0.1957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1747 -0.0133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3316 0.5725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8115 0.8495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4513 -0.2698 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0042 -0.2423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9286 -0.9321 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1118 -1.1141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9369 -1.1312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1713 -0.3399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7763 0.2215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9512 0.2386 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7166 -0.5527 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1342 -0.3836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6487 -0.6507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2750 -1.7777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4829 -1.3809 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6300 -0.8180 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3417 -1.5580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7673 -2.2953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4822 -1.5580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8941 -2.2715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7180 -2.2715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1305 -1.5571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9555 -1.5571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3680 -2.2715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9555 -2.9860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1305 -2.9860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1919 -2.2715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9317 -3.2637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7359 -3.0785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7697 -2.2539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2191 -1.5617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4148 -1.7468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3809 -2.5714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7747 -2.5499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3693 -2.9275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2521 -3.8672 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3568 -3.4316 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3314 -2.6053 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5356 -3.5661 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4835 -3.5661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
14 17 1 0 0 0 0
1 18 1 0 0 0 0
3 19 1 0 0 0 0
8 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
21 29 1 0 0 0 0
22 30 1 0 0 0 0
23 31 1 0 0 0 0
24 19 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
37 38 1 0 0 0 0
38 39 1 0 0 0 0
32 40 1 0 0 0 0
33 41 1 0 0 0 0
34 42 1 0 0 0 0
35 20 1 0 0 0 0
43 44 2 0 0 0 0
43 45 1 0 0 0 0
45 46 2 0 0 0 0
46 47 1 0 0 0 0
47 48 2 0 0 0 0
48 49 1 0 0 0 0
49 50 2 0 0 0 0
50 51 1 0 0 0 0
51 52 2 0 0 0 0
52 47 1 0 0 0 0
50 53 1 0 0 0 0
54 55 1 1 0 0 0
55 56 1 1 0 0 0
57 56 1 1 0 0 0
57 58 1 0 0 0 0
58 59 1 0 0 0 0
59 54 1 0 0 0 0
59 60 1 0 0 0 0
60 61 1 0 0 0 0
54 62 1 0 0 0 0
55 63 1 0 0 0 0
56 64 1 0 0 0 0
57 42 1 0 0 0 0
43 39 1 0 0 0 0
51 65 1 0 0 0 0
65 66 1 0 0 0 0
S SKP 8
ID FL7AAAGL0062
KNApSAcK_ID C00014838
NAME Pelargonidin 3-O-[6-O-(E)-feruloyl-2-O-beta-D-glucopyranosyl-beta-D-glucopyranoside]-5-O-(beta-D-glucopyranoside);Pelargonidin 3-(6-ferulyl-2-glucosylglucoside)-5-glucoside
CAS_RN 185027-87-6
FORMULA C43H49O23
EXACTMASS 933.266462874
AVERAGEMASS 933.83536
SMILES c(O)(c2)cc(c(c4)c([o+1]c(c4OC(C5OC(C(O)7)OC(C(O)C7O)CO)OC(COC(=O)C=Cc(c6)cc(OC)c(c6)O)C(C(O)5)O)c(c3)ccc(O)c3)2)OC(C1O)OC(CO)C(C1O)O
M END