Mol:FL7AAAGO0004
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 41 45 0 0 0 0 0 0 0 0999 V2000 | + | 41 45 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.0960 1.1330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0960 1.1330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0960 0.4906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0960 0.4906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5397 0.1694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5397 0.1694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0167 0.4906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0167 0.4906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0167 1.1330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0167 1.1330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5397 1.4542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5397 1.4542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5730 0.1694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5730 0.1694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1293 0.4906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1293 0.4906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1293 1.1330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1293 1.1330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5730 1.4542 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 0.5730 1.4542 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6854 1.4540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6854 1.4540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2523 1.1267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2523 1.1267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8193 1.4540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8193 1.4540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8193 2.1087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8193 2.1087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2523 2.4361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2523 2.4361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6854 2.1087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6854 2.1087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3861 2.4360 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3861 2.4360 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6521 1.4540 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6521 1.4540 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5397 -0.4727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5397 -0.4727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8852 -0.3405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8852 -0.3405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2385 -0.4431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2385 -0.4431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7229 -1.1238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7229 -1.1238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9804 -0.8350 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9804 -0.8350 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2639 -0.8273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2639 -0.8273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7845 -0.3065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7845 -0.3065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5430 -0.5789 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5430 -0.5789 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8150 -0.7760 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8150 -0.7760 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5267 -1.1629 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5267 -1.1629 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5549 -1.5492 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5549 -1.5492 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7659 -1.5077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7659 -1.5077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3941 -1.8704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3941 -1.8704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1948 -1.1729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1948 -1.1729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3941 -0.4712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3941 -0.4712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7659 -0.1084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7659 -0.1084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9652 -0.8059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9652 -0.8059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7355 -0.8059 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7355 -0.8059 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9272 -2.1099 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9272 -2.1099 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2425 -2.4361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2425 -2.4361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8150 -1.5310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8150 -1.5310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8386 0.2285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8386 0.2285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1241 -0.1840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1241 -0.1840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 31 30 1 1 0 0 0 | + | 31 30 1 1 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 35 30 1 1 0 0 0 | + | 35 30 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 30 37 1 0 0 0 0 | + | 30 37 1 0 0 0 0 |
| − | 31 38 1 0 0 0 0 | + | 31 38 1 0 0 0 0 |
| − | 32 39 1 0 0 0 0 | + | 32 39 1 0 0 0 0 |
| − | 34 20 1 0 0 0 0 | + | 34 20 1 0 0 0 0 |
| − | 26 40 1 0 0 0 0 | + | 26 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 44 -5.5949 4.7454 | + | M SBV 1 44 -5.5949 4.7454 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AAAGS0004 | + | ID FL7AAAGS0004 |
| − | KNApSAcK_ID C00006641 | + | KNApSAcK_ID C00006641 |
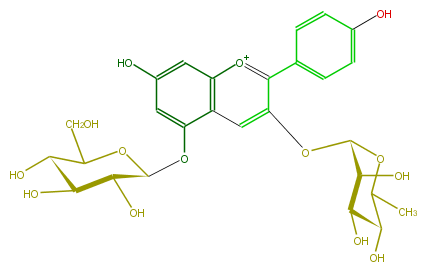
| − | NAME Pelargonidin 3-rhamnoside-5-glucoside | + | NAME Pelargonidin 3-rhamnoside-5-glucoside |
| − | CAS_RN 53925-32-9 | + | CAS_RN 53925-32-9 |
| − | FORMULA C27H31O14 | + | FORMULA C27H31O14 |
| − | EXACTMASS 579.1713807 | + | EXACTMASS 579.1713807 |
| − | AVERAGEMASS 579.52664 | + | AVERAGEMASS 579.52664 |
| − | SMILES O(c(c3)c([o+1]c(c4)c3c(OC(O5)C(C(C(O)C5CO)O)O)cc4O)c(c2)ccc(O)c2)C(O1)C(O)C(O)C(O)C(C)1 | + | SMILES O(c(c3)c([o+1]c(c4)c3c(OC(O5)C(C(C(O)C5CO)O)O)cc4O)c(c2)ccc(O)c2)C(O1)C(O)C(O)C(O)C(C)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
41 45 0 0 0 0 0 0 0 0999 V2000
-1.0960 1.1330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0960 0.4906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5397 0.1694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0167 0.4906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0167 1.1330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5397 1.4542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5730 0.1694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1293 0.4906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1293 1.1330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5730 1.4542 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
1.6854 1.4540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2523 1.1267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8193 1.4540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8193 2.1087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2523 2.4361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6854 2.1087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3861 2.4360 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6521 1.4540 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5397 -0.4727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8852 -0.3405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2385 -0.4431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7229 -1.1238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9804 -0.8350 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2639 -0.8273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7845 -0.3065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5430 -0.5789 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8150 -0.7760 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5267 -1.1629 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5549 -1.5492 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7659 -1.5077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3941 -1.8704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1948 -1.1729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3941 -0.4712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7659 -0.1084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9652 -0.8059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7355 -0.8059 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9272 -2.1099 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2425 -2.4361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8150 -1.5310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8386 0.2285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1241 -0.1840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
14 17 1 0 0 0 0
1 18 1 0 0 0 0
3 19 1 0 0 0 0
8 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 19 1 0 0 0 0
31 30 1 1 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 1 0 0 0
35 30 1 1 0 0 0
35 36 1 0 0 0 0
30 37 1 0 0 0 0
31 38 1 0 0 0 0
32 39 1 0 0 0 0
34 20 1 0 0 0 0
26 40 1 0 0 0 0
40 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 44
M SMT 1 ^CH2OH
M SBV 1 44 -5.5949 4.7454
S SKP 8
ID FL7AAAGS0004
KNApSAcK_ID C00006641
NAME Pelargonidin 3-rhamnoside-5-glucoside
CAS_RN 53925-32-9
FORMULA C27H31O14
EXACTMASS 579.1713807
AVERAGEMASS 579.52664
SMILES O(c(c3)c([o+1]c(c4)c3c(OC(O5)C(C(C(O)C5CO)O)O)cc4O)c(c2)ccc(O)c2)C(O1)C(O)C(O)C(O)C(C)1
M END