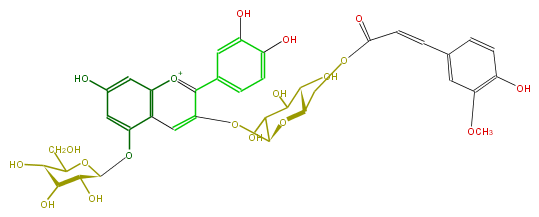
Mol:FL7AACGL0052
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 56 61 0 0 0 0 0 0 0 0999 V2000 | + | 56 61 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.0001 0.8204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0001 0.8204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0001 0.1780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0001 0.1780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4438 -0.1431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4438 -0.1431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8875 0.1780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8875 0.1780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8875 0.8204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8875 0.8204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4438 1.1416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4438 1.1416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3312 -0.1431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3312 -0.1431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2251 0.1780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2251 0.1780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2251 0.8204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2251 0.8204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3312 1.1416 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -0.3312 1.1416 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7812 1.1415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7812 1.1415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3482 0.8141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3482 0.8141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9151 1.1415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9151 1.1415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9151 1.7961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9151 1.7961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3482 2.1235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3482 2.1235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7812 1.7961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7812 1.7961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5562 1.1415 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5562 1.1415 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4819 2.1234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4819 2.1234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4438 -0.7853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4438 -0.7853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2099 0.0044 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2099 0.0044 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3482 2.7780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3482 2.7780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6026 -1.0595 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -3.6026 -1.0595 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -3.2314 -1.5494 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.2314 -1.5494 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.6969 -1.3416 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.6969 -1.3416 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.1812 -1.3360 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.1812 -1.3360 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.5559 -0.9611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5559 -0.9611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0235 -1.2079 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -3.0235 -1.2079 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -4.1935 -1.0077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1935 -1.0077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5817 -2.0106 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5817 -2.0106 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3907 -1.8557 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3907 -1.8557 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3942 -0.6328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3942 -0.6328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9720 0.1487 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.9720 0.1487 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.0766 -0.4438 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.0766 -0.4438 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.4134 0.0547 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4134 0.0547 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9655 0.3021 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.9655 0.3021 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.8611 0.8947 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.8611 0.8947 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.5241 0.3962 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.5241 0.3962 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.6403 -0.3251 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6403 -0.3251 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2509 0.7864 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2509 0.7864 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5210 1.2024 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5210 1.2024 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2088 0.8140 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2088 0.8140 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2724 0.0579 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2724 0.0579 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9591 1.5963 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9591 1.5963 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5935 2.0744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5935 2.0744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2941 2.3109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2941 2.3109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9504 2.0169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9504 2.0169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.5764 1.7440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5764 1.7440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.6337 1.0850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.6337 1.0850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.2334 0.8053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.2334 0.8053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.7754 1.1848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.7754 1.1848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.7179 1.8438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.7179 1.8438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.1183 2.1234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.1183 2.1234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.3747 0.9053 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.3747 0.9053 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3245 2.6484 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3245 2.6484 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.1463 -0.1909 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.1463 -0.1909 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.0594 -1.1869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.0594 -1.1869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 27 31 1 0 0 0 0 | + | 27 31 1 0 0 0 0 |
| − | 25 19 1 0 0 0 0 | + | 25 19 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 37 39 1 0 0 0 0 | + | 37 39 1 0 0 0 0 |
| − | 36 40 1 0 0 0 0 | + | 36 40 1 0 0 0 0 |
| − | 35 41 1 0 0 0 0 | + | 35 41 1 0 0 0 0 |
| − | 33 20 1 0 0 0 0 | + | 33 20 1 0 0 0 0 |
| − | 31 42 1 0 0 0 0 | + | 31 42 1 0 0 0 0 |
| − | 41 43 1 0 0 0 0 | + | 41 43 1 0 0 0 0 |
| − | 45 46 2 0 0 0 0 | + | 45 46 2 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 2 0 0 0 0 | + | 47 48 2 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 51 52 2 0 0 0 0 | + | 51 52 2 0 0 0 0 |
| − | 52 47 1 0 0 0 0 | + | 52 47 1 0 0 0 0 |
| − | 50 53 1 0 0 0 0 | + | 50 53 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 44 54 2 0 0 0 0 | + | 44 54 2 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 49 55 1 0 0 0 0 | + | 49 55 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 31 42 | + | M SAL 2 2 31 42 |
| − | M SBL 2 1 33 | + | M SBL 2 1 33 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 33 -3.4884 -0.7059 | + | M SVB 2 33 -3.4884 -0.7059 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 55 56 | + | M SAL 1 2 55 56 |
| − | M SBL 1 1 60 | + | M SBL 1 1 60 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 60 0.4906 -0.481 | + | M SVB 1 60 0.4906 -0.481 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AACGL0052 | + | ID FL7AACGL0052 |
| − | KNApSAcK_ID C00006823 | + | KNApSAcK_ID C00006823 |
| − | NAME Cyanidin 3-(6''-ferulylglucoside)-5-glucoside | + | NAME Cyanidin 3-(6''-ferulylglucoside)-5-glucoside |
| − | CAS_RN 79410-82-5 | + | CAS_RN 79410-82-5 |
| − | FORMULA C37H39O19 | + | FORMULA C37H39O19 |
| − | EXACTMASS 787.208554066 | + | EXACTMASS 787.208554066 |
| − | AVERAGEMASS 787.69416 | + | AVERAGEMASS 787.69416 |
| − | SMILES C(O)C(O1)[C@H](O)[C@H](O)[C@H](O)[C@H](Oc(c6)c(c(cc(O)6)2)cc(O[C@@H](C(O)5)O[C@@H]([C@H](O)C(O)5)COC(=O)C=Cc(c4)ccc(O)c(OC)4)c(c(c3)ccc(c(O)3)O)[o+1]2)1 | + | SMILES C(O)C(O1)[C@H](O)[C@H](O)[C@H](O)[C@H](Oc(c6)c(c(cc(O)6)2)cc(O[C@@H](C(O)5)O[C@@H]([C@H](O)C(O)5)COC(=O)C=Cc(c4)ccc(O)c(OC)4)c(c(c3)ccc(c(O)3)O)[o+1]2)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
56 61 0 0 0 0 0 0 0 0999 V2000
-2.0001 0.8204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0001 0.1780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4438 -0.1431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8875 0.1780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8875 0.8204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4438 1.1416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3312 -0.1431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2251 0.1780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2251 0.8204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3312 1.1416 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
0.7812 1.1415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3482 0.8141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9151 1.1415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9151 1.7961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3482 2.1235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7812 1.7961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5562 1.1415 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4819 2.1234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4438 -0.7853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2099 0.0044 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3482 2.7780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6026 -1.0595 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-3.2314 -1.5494 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.6969 -1.3416 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.1812 -1.3360 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.5559 -0.9611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0235 -1.2079 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-4.1935 -1.0077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5817 -2.0106 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3907 -1.8557 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3942 -0.6328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9720 0.1487 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.0766 -0.4438 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.4134 0.0547 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9655 0.3021 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.8611 0.8947 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.5241 0.3962 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.6403 -0.3251 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2509 0.7864 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5210 1.2024 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2088 0.8140 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2724 0.0579 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9591 1.5963 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5935 2.0744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2941 2.3109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9504 2.0169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.5764 1.7440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.6337 1.0850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.2334 0.8053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.7754 1.1848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.7179 1.8438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.1183 2.1234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.3747 0.9053 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3245 2.6484 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.1463 -0.1909 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.0594 -1.1869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
15 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
27 31 1 0 0 0 0
25 19 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
32 38 1 0 0 0 0
37 39 1 0 0 0 0
36 40 1 0 0 0 0
35 41 1 0 0 0 0
33 20 1 0 0 0 0
31 42 1 0 0 0 0
41 43 1 0 0 0 0
45 46 2 0 0 0 0
46 47 1 0 0 0 0
47 48 2 0 0 0 0
48 49 1 0 0 0 0
49 50 2 0 0 0 0
50 51 1 0 0 0 0
51 52 2 0 0 0 0
52 47 1 0 0 0 0
50 53 1 0 0 0 0
44 45 1 0 0 0 0
44 54 2 0 0 0 0
43 44 1 0 0 0 0
49 55 1 0 0 0 0
55 56 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 31 42
M SBL 2 1 33
M SMT 2 CH2OH
M SVB 2 33 -3.4884 -0.7059
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 55 56
M SBL 1 1 60
M SMT 1 OCH3
M SVB 1 60 0.4906 -0.481
S SKP 8
ID FL7AACGL0052
KNApSAcK_ID C00006823
NAME Cyanidin 3-(6''-ferulylglucoside)-5-glucoside
CAS_RN 79410-82-5
FORMULA C37H39O19
EXACTMASS 787.208554066
AVERAGEMASS 787.69416
SMILES C(O)C(O1)[C@H](O)[C@H](O)[C@H](O)[C@H](Oc(c6)c(c(cc(O)6)2)cc(O[C@@H](C(O)5)O[C@@H]([C@H](O)C(O)5)COC(=O)C=Cc(c4)ccc(O)c(OC)4)c(c(c3)ccc(c(O)3)O)[o+1]2)1
M END