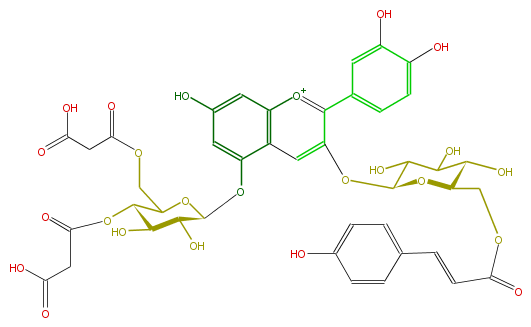
Mol:FL7AACGL0056
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 66 71 0 0 0 0 0 0 0 0999 V2000 | + | 66 71 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.1339 1.0426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1339 1.0426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1339 0.4002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1339 0.4002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5776 0.0791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5776 0.0791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0213 0.4002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0213 0.4002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0213 1.0426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0213 1.0426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5776 1.3638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5776 1.3638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5350 0.0791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5350 0.0791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0913 0.4002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0913 0.4002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0913 1.0426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0913 1.0426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5350 1.3638 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 0.5350 1.3638 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6474 1.3637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6474 1.3637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2143 1.0363 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2143 1.0363 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7813 1.3637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7813 1.3637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7813 2.0183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7813 2.0183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2143 2.3457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2143 2.3457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6474 2.0183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6474 2.0183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6900 1.3637 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6900 1.3637 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3481 2.3456 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3481 2.3456 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5776 -0.5631 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5776 -0.5631 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5074 -0.3205 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5074 -0.3205 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2143 3.0002 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2143 3.0002 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7364 -0.8373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7364 -0.8373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3652 -1.3272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3652 -1.3272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8307 -1.1194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8307 -1.1194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3150 -1.1138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3150 -1.1138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6897 -0.7389 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6897 -0.7389 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1574 -0.9857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1574 -0.9857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2502 -1.1339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2502 -1.1339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9438 -1.3554 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9438 -1.3554 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5245 -1.6335 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5245 -1.6335 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6222 -0.4837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6222 -0.4837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7679 0.0209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7679 0.0209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4671 -0.5002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4671 -0.5002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0456 -0.3348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0456 -0.3348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6276 -0.5002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6276 -0.5002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9284 0.0209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9284 0.0209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3499 -0.1444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3499 -0.1444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2092 -0.1288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2092 -0.1288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5881 0.2681 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5881 0.2681 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6318 -0.1676 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6318 -0.1676 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1943 -0.5043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1943 -0.5043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6222 0.2177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6222 0.2177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5862 -1.5149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5862 -1.5149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4075 -2.2890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4075 -2.2890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6765 -2.4003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6765 -2.4003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3075 -1.7830 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3075 -1.7830 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6248 -1.8002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6248 -1.8002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2940 -1.2272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2940 -1.2272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6323 -1.2272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6323 -1.2272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3015 -1.8002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3015 -1.8002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6323 -2.3732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6323 -2.3732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2940 -2.3732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2940 -2.3732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6401 -1.8002 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6401 -1.8002 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9826 -2.5558 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9826 -2.5558 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1661 0.5317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1661 0.5317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6064 0.2775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6064 0.2775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0591 0.5388 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0591 0.5388 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5646 0.2469 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5646 0.2469 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1661 1.1587 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1661 1.1587 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0591 1.1218 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0591 1.1218 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9983 -1.3344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9983 -1.3344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4845 -1.0537 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4845 -1.0537 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9983 -2.0718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9983 -2.0718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4901 -2.3557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4901 -2.3557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4901 -3.0002 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4901 -3.0002 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9826 -2.0714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9826 -2.0714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 27 31 1 0 0 0 0 | + | 27 31 1 0 0 0 0 |
| − | 25 19 1 0 0 0 0 | + | 25 19 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 37 39 1 0 0 0 0 | + | 37 39 1 0 0 0 0 |
| − | 36 40 1 0 0 0 0 | + | 36 40 1 0 0 0 0 |
| − | 35 41 1 0 0 0 0 | + | 35 41 1 0 0 0 0 |
| − | 33 20 1 0 0 0 0 | + | 33 20 1 0 0 0 0 |
| − | 31 42 1 0 0 0 0 | + | 31 42 1 0 0 0 0 |
| − | 41 43 1 0 0 0 0 | + | 41 43 1 0 0 0 0 |
| − | 45 46 2 0 0 0 0 | + | 45 46 2 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 2 0 0 0 0 | + | 47 48 2 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 51 52 2 0 0 0 0 | + | 51 52 2 0 0 0 0 |
| − | 52 47 1 0 0 0 0 | + | 52 47 1 0 0 0 0 |
| − | 50 53 1 0 0 0 0 | + | 50 53 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 44 54 2 0 0 0 0 | + | 44 54 2 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 42 55 1 0 0 0 0 | + | 42 55 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 56 57 1 0 0 0 0 | + | 56 57 1 0 0 0 0 |
| − | 57 58 2 0 0 0 0 | + | 57 58 2 0 0 0 0 |
| − | 55 59 2 0 0 0 0 | + | 55 59 2 0 0 0 0 |
| − | 57 60 1 0 0 0 0 | + | 57 60 1 0 0 0 0 |
| − | 28 61 1 0 0 0 0 | + | 28 61 1 0 0 0 0 |
| − | 61 62 2 0 0 0 0 | + | 61 62 2 0 0 0 0 |
| − | 61 63 1 0 0 0 0 | + | 61 63 1 0 0 0 0 |
| − | 63 64 1 0 0 0 0 | + | 63 64 1 0 0 0 0 |
| − | 64 65 2 0 0 0 0 | + | 64 65 2 0 0 0 0 |
| − | 64 66 1 0 0 0 0 | + | 64 66 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AACGL0056 | + | ID FL7AACGL0056 |
| − | KNApSAcK_ID C00006827 | + | KNApSAcK_ID C00006827 |
| − | NAME Cyanidin 3-(6''-p-coumarylglucoside)-5-4''',6'''-dimalonylglucoside) | + | NAME Cyanidin 3-(6''-p-coumarylglucoside)-5-4''',6'''-dimalonylglucoside) |
| − | CAS_RN - | + | CAS_RN - |
| − | FORMULA C42H41O24 | + | FORMULA C42H41O24 |
| − | EXACTMASS 929.19877724 | + | EXACTMASS 929.19877724 |
| − | AVERAGEMASS 929.76054 | + | AVERAGEMASS 929.76054 |
| − | SMILES O(C(Oc(c6)c(c(cc6O)4)cc(c(c(c5)ccc(c5O)O)[o+1]4)OC(O2)C(O)C(C(C2COC(C=Cc(c3)ccc(O)c3)=O)O)O)1)C(COC(=O)CC(O)=O)C(C(O)C1O)OC(=O)CC(O)=O | + | SMILES O(C(Oc(c6)c(c(cc6O)4)cc(c(c(c5)ccc(c5O)O)[o+1]4)OC(O2)C(O)C(C(C2COC(C=Cc(c3)ccc(O)c3)=O)O)O)1)C(COC(=O)CC(O)=O)C(C(O)C1O)OC(=O)CC(O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
66 71 0 0 0 0 0 0 0 0999 V2000
-1.1339 1.0426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1339 0.4002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5776 0.0791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0213 0.4002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0213 1.0426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5776 1.3638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5350 0.0791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0913 0.4002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0913 1.0426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5350 1.3638 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
1.6474 1.3637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2143 1.0363 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7813 1.3637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7813 2.0183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2143 2.3457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6474 2.0183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6900 1.3637 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3481 2.3456 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5776 -0.5631 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5074 -0.3205 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2143 3.0002 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7364 -0.8373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3652 -1.3272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8307 -1.1194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3150 -1.1138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6897 -0.7389 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1574 -0.9857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2502 -1.1339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9438 -1.3554 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5245 -1.6335 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6222 -0.4837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7679 0.0209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4671 -0.5002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0456 -0.3348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6276 -0.5002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9284 0.0209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3499 -0.1444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2092 -0.1288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5881 0.2681 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6318 -0.1676 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1943 -0.5043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6222 0.2177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5862 -1.5149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4075 -2.2890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6765 -2.4003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3075 -1.7830 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6248 -1.8002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2940 -1.2272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6323 -1.2272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3015 -1.8002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6323 -2.3732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2940 -2.3732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6401 -1.8002 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9826 -2.5558 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1661 0.5317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6064 0.2775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0591 0.5388 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5646 0.2469 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1661 1.1587 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0591 1.1218 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9983 -1.3344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4845 -1.0537 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9983 -2.0718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4901 -2.3557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4901 -3.0002 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9826 -2.0714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
15 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
27 31 1 0 0 0 0
25 19 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
32 38 1 0 0 0 0
37 39 1 0 0 0 0
36 40 1 0 0 0 0
35 41 1 0 0 0 0
33 20 1 0 0 0 0
31 42 1 0 0 0 0
41 43 1 0 0 0 0
45 46 2 0 0 0 0
46 47 1 0 0 0 0
47 48 2 0 0 0 0
48 49 1 0 0 0 0
49 50 2 0 0 0 0
50 51 1 0 0 0 0
51 52 2 0 0 0 0
52 47 1 0 0 0 0
50 53 1 0 0 0 0
44 45 1 0 0 0 0
44 54 2 0 0 0 0
43 44 1 0 0 0 0
42 55 1 0 0 0 0
55 56 1 0 0 0 0
56 57 1 0 0 0 0
57 58 2 0 0 0 0
55 59 2 0 0 0 0
57 60 1 0 0 0 0
28 61 1 0 0 0 0
61 62 2 0 0 0 0
61 63 1 0 0 0 0
63 64 1 0 0 0 0
64 65 2 0 0 0 0
64 66 1 0 0 0 0
S SKP 8
ID FL7AACGL0056
KNApSAcK_ID C00006827
NAME Cyanidin 3-(6''-p-coumarylglucoside)-5-4''',6'''-dimalonylglucoside)
CAS_RN -
FORMULA C42H41O24
EXACTMASS 929.19877724
AVERAGEMASS 929.76054
SMILES O(C(Oc(c6)c(c(cc6O)4)cc(c(c(c5)ccc(c5O)O)[o+1]4)OC(O2)C(O)C(C(C2COC(C=Cc(c3)ccc(O)c3)=O)O)O)1)C(COC(=O)CC(O)=O)C(C(O)C1O)OC(=O)CC(O)=O
M END