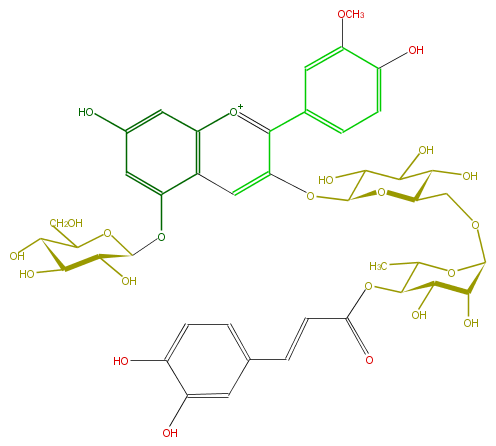
Mol:FL7AADGL0026
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 66 72 0 0 0 0 0 0 0 0999 V2000 | + | 66 72 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.4470 1.2302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4470 1.2302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4469 0.4004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4469 0.4004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7283 -0.0147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7283 -0.0147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0094 0.4004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0094 0.4004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0095 1.2303 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0095 1.2303 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7282 1.6452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7282 1.6452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2907 -0.0147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2907 -0.0147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4281 0.4004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4281 0.4004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4281 1.2302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4281 1.2302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2907 1.6452 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -0.2907 1.6452 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1465 1.6451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1465 1.6451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8791 1.2221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8791 1.2221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6116 1.6451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6116 1.6451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6116 2.4910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6116 2.4910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8790 2.9138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8790 2.9138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1466 2.4910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1466 2.4910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1654 1.6450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1654 1.6450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2830 2.8786 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2830 2.8786 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7282 -0.8442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7282 -0.8442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2517 -0.0426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2517 -0.0426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4099 -1.3835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4099 -1.3835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0634 -1.9838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0634 -1.9838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7298 -1.7933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7298 -1.7933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4003 -1.9838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4003 -1.9838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7469 -1.3835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7469 -1.3835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0804 -1.5739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0804 -1.5739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7234 -1.4251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7234 -1.4251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3452 0.4629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3452 0.4629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9986 -0.1374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9986 -0.1374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6652 0.0530 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6652 0.0530 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3355 -0.1373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3355 -0.1373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6822 0.4629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6822 0.4629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0156 0.2725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0156 0.2725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6281 0.2934 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6281 0.2934 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4980 0.8797 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4980 0.8797 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3818 0.3585 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3818 0.3585 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9723 0.0057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9723 0.0057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5584 -0.6140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5584 -0.6140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4065 -1.8309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4065 -1.8309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3458 -2.4083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3458 -2.4083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4003 -2.5685 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4003 -2.5685 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1574 -0.8943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1574 -0.8943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6776 -1.5271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6776 -1.5271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9870 -1.2586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9870 -1.2586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3206 -1.2515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3206 -1.2515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8049 -0.7673 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8049 -0.7673 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4092 -1.0862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4092 -1.0862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6926 -1.2414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6926 -1.2414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3544 -1.5898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3544 -1.5898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4441 -1.7400 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4441 -1.7400 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9624 -2.4933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9624 -2.4933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4331 -3.3086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4331 -3.3086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1665 -2.4969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1665 -2.4969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7706 -3.3264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7706 -3.3264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0215 -3.3298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0215 -3.3298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3936 -2.6205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3936 -2.6205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2236 -2.6243 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2236 -2.6243 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6386 -3.3373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6386 -3.3373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2236 -4.0468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2236 -4.0468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3935 -4.0429 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3935 -4.0429 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4679 -3.3412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4679 -3.3412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6346 -4.7674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6346 -4.7674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8790 3.6081 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8790 3.6081 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4495 4.7674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4495 4.7674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9933 -0.5725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9933 -0.5725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7469 0.1812 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7469 0.1812 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 1 0 0 0 | + | 29 30 1 1 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 32 31 1 1 0 0 0 | + | 32 31 1 1 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 28 1 0 0 0 0 | + | 33 28 1 0 0 0 0 |
| − | 28 34 1 0 0 0 0 | + | 28 34 1 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 32 36 1 0 0 0 0 | + | 32 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 25 38 1 0 0 0 0 | + | 25 38 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 22 39 1 0 0 0 0 | + | 22 39 1 0 0 0 0 |
| − | 23 40 1 0 0 0 0 | + | 23 40 1 0 0 0 0 |
| − | 24 41 1 0 0 0 0 | + | 24 41 1 0 0 0 0 |
| − | 29 20 1 0 0 0 0 | + | 29 20 1 0 0 0 0 |
| − | 42 43 1 1 0 0 0 | + | 42 43 1 1 0 0 0 |
| − | 43 44 1 1 0 0 0 | + | 43 44 1 1 0 0 0 |
| − | 45 44 1 1 0 0 0 | + | 45 44 1 1 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 42 1 0 0 0 0 | + | 47 42 1 0 0 0 0 |
| − | 42 48 1 0 0 0 0 | + | 42 48 1 0 0 0 0 |
| − | 43 49 1 0 0 0 0 | + | 43 49 1 0 0 0 0 |
| − | 44 50 1 0 0 0 0 | + | 44 50 1 0 0 0 0 |
| − | 45 19 1 0 0 0 0 | + | 45 19 1 0 0 0 0 |
| − | 51 52 2 0 0 0 0 | + | 51 52 2 0 0 0 0 |
| − | 51 53 1 0 0 0 0 | + | 51 53 1 0 0 0 0 |
| − | 53 54 2 0 0 0 0 | + | 53 54 2 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 55 56 2 0 0 0 0 | + | 55 56 2 0 0 0 0 |
| − | 56 57 1 0 0 0 0 | + | 56 57 1 0 0 0 0 |
| − | 57 58 2 0 0 0 0 | + | 57 58 2 0 0 0 0 |
| − | 58 59 1 0 0 0 0 | + | 58 59 1 0 0 0 0 |
| − | 59 60 2 0 0 0 0 | + | 59 60 2 0 0 0 0 |
| − | 60 55 1 0 0 0 0 | + | 60 55 1 0 0 0 0 |
| − | 58 61 1 0 0 0 0 | + | 58 61 1 0 0 0 0 |
| − | 39 51 1 0 0 0 0 | + | 39 51 1 0 0 0 0 |
| − | 59 62 1 0 0 0 0 | + | 59 62 1 0 0 0 0 |
| − | 63 64 1 0 0 0 0 | + | 63 64 1 0 0 0 0 |
| − | 15 63 1 0 0 0 0 | + | 15 63 1 0 0 0 0 |
| − | 65 66 1 0 0 0 0 | + | 65 66 1 0 0 0 0 |
| − | 47 65 1 0 0 0 0 | + | 47 65 1 0 0 0 0 |
| − | M CHG 1 10 1 | + | M CHG 1 10 1 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 63 64 | + | M SAL 1 2 63 64 |
| − | M SBL 1 1 70 | + | M SBL 1 1 70 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 70 0.0000 -0.6943 | + | M SBV 1 70 0.0000 -0.6943 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 65 66 | + | M SAL 2 2 65 66 |
| − | M SBL 2 1 72 | + | M SBL 2 1 72 |
| − | M SMT 2 ^ CH2OH | + | M SMT 2 ^ CH2OH |
| − | M SBV 2 72 0.5841 -0.5138 | + | M SBV 2 72 0.5841 -0.5138 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL7AADGL0026 | + | ID FL7AADGL0026 |
| − | FORMULA C43H49O23 | + | FORMULA C43H49O23 |
| − | EXACTMASS 933.266462874 | + | EXACTMASS 933.266462874 |
| − | AVERAGEMASS 933.83536 | + | AVERAGEMASS 933.83536 |
| − | SMILES c(O)(c7)c(O)cc(c7)C=CC(=O)OC(C(C)1)C(C(O)C(OCC(O2)C(O)C(C(C(Oc(c(c(c6)cc(c(c6)O)OC)4)cc(c3OC(O5)C(C(O)C(O)C5CO)O)c([o+1]4)cc(c3)O)2)O)O)O1)O | + | SMILES c(O)(c7)c(O)cc(c7)C=CC(=O)OC(C(C)1)C(C(O)C(OCC(O2)C(O)C(C(C(Oc(c(c(c6)cc(c(c6)O)OC)4)cc(c3OC(O5)C(C(O)C(O)C5CO)O)c([o+1]4)cc(c3)O)2)O)O)O1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
66 72 0 0 0 0 0 0 0 0999 V2000
-2.4470 1.2302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4469 0.4004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7283 -0.0147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0094 0.4004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0095 1.2303 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7282 1.6452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2907 -0.0147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4281 0.4004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4281 1.2302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2907 1.6452 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
1.1465 1.6451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8791 1.2221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6116 1.6451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6116 2.4910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8790 2.9138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1466 2.4910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1654 1.6450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2830 2.8786 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7282 -0.8442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2517 -0.0426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4099 -1.3835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0634 -1.9838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7298 -1.7933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4003 -1.9838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7469 -1.3835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0804 -1.5739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7234 -1.4251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3452 0.4629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9986 -0.1374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6652 0.0530 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3355 -0.1373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6822 0.4629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0156 0.2725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6281 0.2934 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4980 0.8797 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3818 0.3585 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9723 0.0057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5584 -0.6140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4065 -1.8309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3458 -2.4083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4003 -2.5685 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1574 -0.8943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6776 -1.5271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9870 -1.2586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3206 -1.2515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8049 -0.7673 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4092 -1.0862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6926 -1.2414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3544 -1.5898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4441 -1.7400 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9624 -2.4933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4331 -3.3086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1665 -2.4969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7706 -3.3264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0215 -3.3298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3936 -2.6205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2236 -2.6243 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6386 -3.3373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2236 -4.0468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3935 -4.0429 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4679 -3.3412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6346 -4.7674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8790 3.6081 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4495 4.7674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9933 -0.5725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7469 0.1812 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
28 29 1 0 0 0 0
29 30 1 1 0 0 0
30 31 1 1 0 0 0
32 31 1 1 0 0 0
32 33 1 0 0 0 0
33 28 1 0 0 0 0
28 34 1 0 0 0 0
33 35 1 0 0 0 0
32 36 1 0 0 0 0
31 37 1 0 0 0 0
25 38 1 0 0 0 0
37 38 1 0 0 0 0
22 39 1 0 0 0 0
23 40 1 0 0 0 0
24 41 1 0 0 0 0
29 20 1 0 0 0 0
42 43 1 1 0 0 0
43 44 1 1 0 0 0
45 44 1 1 0 0 0
45 46 1 0 0 0 0
46 47 1 0 0 0 0
47 42 1 0 0 0 0
42 48 1 0 0 0 0
43 49 1 0 0 0 0
44 50 1 0 0 0 0
45 19 1 0 0 0 0
51 52 2 0 0 0 0
51 53 1 0 0 0 0
53 54 2 0 0 0 0
54 55 1 0 0 0 0
55 56 2 0 0 0 0
56 57 1 0 0 0 0
57 58 2 0 0 0 0
58 59 1 0 0 0 0
59 60 2 0 0 0 0
60 55 1 0 0 0 0
58 61 1 0 0 0 0
39 51 1 0 0 0 0
59 62 1 0 0 0 0
63 64 1 0 0 0 0
15 63 1 0 0 0 0
65 66 1 0 0 0 0
47 65 1 0 0 0 0
M CHG 1 10 1
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 63 64
M SBL 1 1 70
M SMT 1 OCH3
M SBV 1 70 0.0000 -0.6943
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 65 66
M SBL 2 1 72
M SMT 2 ^ CH2OH
M SBV 2 72 0.5841 -0.5138
S SKP 5
ID FL7AADGL0026
FORMULA C43H49O23
EXACTMASS 933.266462874
AVERAGEMASS 933.83536
SMILES c(O)(c7)c(O)cc(c7)C=CC(=O)OC(C(C)1)C(C(O)C(OCC(O2)C(O)C(C(C(Oc(c(c(c6)cc(c(c6)O)OC)4)cc(c3OC(O5)C(C(O)C(O)C5CO)O)c([o+1]4)cc(c3)O)2)O)O)O1)O
M END