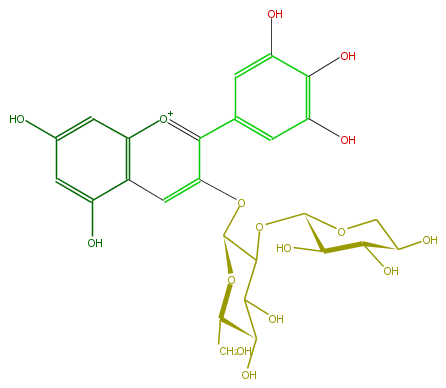
Mol:FL7AAGGL0003
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.3458 1.4341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3458 1.4341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3458 0.6092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3458 0.6092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6314 0.1967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6314 0.1967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9169 0.6092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9169 0.6092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9169 1.4341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9169 1.4341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6314 1.8466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6314 1.8466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2024 0.1967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2024 0.1967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4880 0.6092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4880 0.6092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4880 1.4341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4880 1.4341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2024 1.8466 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -1.2024 1.8466 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2262 1.8465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2262 1.8465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9544 1.4262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9544 1.4262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6826 1.8465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6826 1.8465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6826 2.6873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6826 2.6873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9544 3.1077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9544 3.1077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2262 2.6873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2262 2.6873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0600 1.8465 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0600 1.8465 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4106 3.1076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4106 3.1076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6314 -0.6280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6314 -0.6280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3572 0.1772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3572 0.1772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9544 3.9483 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9544 3.9483 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4106 1.4263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4106 1.4263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6403 -0.9935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6403 -0.9935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0108 -0.4957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0108 -0.4957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1576 -1.3703 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1576 -1.3703 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0668 -2.1601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0668 -2.1601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6403 -2.5685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6403 -2.5685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4161 -1.7833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4161 -1.7833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7217 -0.3100 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7217 -0.3100 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9789 -2.1505 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9789 -2.1505 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4399 -3.2709 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4399 -3.2709 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6012 -0.0596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6012 -0.0596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9910 -0.8198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9910 -0.8198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7726 -0.6644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7726 -0.6644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4800 -0.7813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4800 -0.7813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0575 -0.1776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0575 -0.1776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3578 -0.4027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3578 -0.4027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2794 -0.7253 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2794 -0.7253 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2755 -1.1959 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2755 -1.1959 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0600 -0.5764 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0600 -0.5764 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1168 -2.7835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1168 -2.7835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6580 -3.9483 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6580 -3.9483 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 13 22 1 0 0 0 0 | + | 13 22 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 28 30 1 0 0 0 0 | + | 28 30 1 0 0 0 0 |
| − | 27 31 1 0 0 0 0 | + | 27 31 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 35 40 1 0 0 0 0 | + | 35 40 1 0 0 0 0 |
| − | 29 32 1 0 0 0 0 | + | 29 32 1 0 0 0 0 |
| − | 24 20 1 0 0 0 0 | + | 24 20 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 26 41 1 0 0 0 0 | + | 26 41 1 0 0 0 0 |
| − | M CHG 1 10 1 | + | M CHG 1 10 1 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 46 | + | M SBL 1 1 46 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 46 0.0500 0.6234 | + | M SBV 1 46 0.0500 0.6234 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL7AAGGL0003 | + | ID FL7AAGGL0003 |
| − | FORMULA C26H29O16 | + | FORMULA C26H29O16 |
| − | EXACTMASS 597.1455598800001 | + | EXACTMASS 597.1455598800001 |
| − | AVERAGEMASS 597.4988599999999 | + | AVERAGEMASS 597.4988599999999 |
| − | SMILES C(C(O)1)(C(OC(C5O)OCC(C5O)O)C(Oc(c2)c(c(c4)cc(O)c(c(O)4)O)[o+1]c(c3)c(c(cc3O)O)2)OC1CO)O | + | SMILES C(C(O)1)(C(OC(C5O)OCC(C5O)O)C(Oc(c2)c(c(c4)cc(O)c(c(O)4)O)[o+1]c(c3)c(c(cc3O)O)2)OC1CO)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-3.3458 1.4341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3458 0.6092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6314 0.1967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9169 0.6092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9169 1.4341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6314 1.8466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2024 0.1967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4880 0.6092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4880 1.4341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2024 1.8466 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
0.2262 1.8465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9544 1.4262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6826 1.8465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6826 2.6873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9544 3.1077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2262 2.6873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0600 1.8465 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4106 3.1076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6314 -0.6280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3572 0.1772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9544 3.9483 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4106 1.4263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6403 -0.9935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0108 -0.4957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1576 -1.3703 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0668 -2.1601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6403 -2.5685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4161 -1.7833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7217 -0.3100 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9789 -2.1505 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4399 -3.2709 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6012 -0.0596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9910 -0.8198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7726 -0.6644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4800 -0.7813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0575 -0.1776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3578 -0.4027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2794 -0.7253 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2755 -1.1959 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0600 -0.5764 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1168 -2.7835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6580 -3.9483 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
15 21 1 0 0 0 0
13 22 1 0 0 0 0
23 24 1 0 0 0 0
24 25 1 1 0 0 0
25 26 1 1 0 0 0
27 26 1 1 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
23 29 1 0 0 0 0
28 30 1 0 0 0 0
27 31 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
33 38 1 0 0 0 0
34 39 1 0 0 0 0
35 40 1 0 0 0 0
29 32 1 0 0 0 0
24 20 1 0 0 0 0
41 42 1 0 0 0 0
26 41 1 0 0 0 0
M CHG 1 10 1
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 46
M SMT 1 ^ CH2OH
M SBV 1 46 0.0500 0.6234
S SKP 5
ID FL7AAGGL0003
FORMULA C26H29O16
EXACTMASS 597.1455598800001
AVERAGEMASS 597.4988599999999
SMILES C(C(O)1)(C(OC(C5O)OCC(C5O)O)C(Oc(c2)c(c(c4)cc(O)c(c(O)4)O)[o+1]c(c3)c(c(cc3O)O)2)OC1CO)O
M END