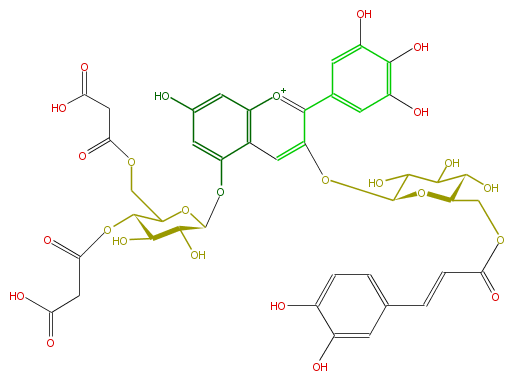
Mol:FL7AAGGL0026
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 68 73 0 0 0 0 0 0 0 0999 V2000 | + | 68 73 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.3512 1.5696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3512 1.5696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3512 0.9273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3512 0.9273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7949 0.6061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7949 0.6061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2386 0.9273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2386 0.9273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2386 1.5696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2386 1.5696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7949 1.8908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7949 1.8908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3177 0.6061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3177 0.6061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8740 0.9273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8740 0.9273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8740 1.5696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8740 1.5696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3177 1.8908 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 0.3177 1.8908 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4301 1.8907 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4301 1.8907 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9970 1.5633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9970 1.5633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5640 1.8907 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5640 1.8907 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5640 2.5454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5640 2.5454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9970 2.8727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9970 2.8727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4301 2.5454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4301 2.5454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9073 1.8907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9073 1.8907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1308 2.8726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1308 2.8726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7949 -0.0360 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7949 -0.0360 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2901 0.2066 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2901 0.2066 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9970 3.5272 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9970 3.5272 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1300 1.5639 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1300 1.5639 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9425 0.3004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9425 0.3004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6417 -0.2206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6417 -0.2206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2202 -0.0553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2202 -0.0553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8021 -0.2206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8021 -0.2206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1030 0.3004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1030 0.3004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5245 0.1351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5245 0.1351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3838 0.1507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3838 0.1507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7627 0.5477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7627 0.5477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5371 0.0498 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5371 0.0498 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3537 -0.2206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3537 -0.2206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8004 -0.9943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8004 -0.9943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4184 -1.6559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4184 -1.6559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6988 -2.1416 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6988 -2.1416 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6552 -1.6559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6552 -1.6559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2737 -2.3168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2737 -2.3168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5119 -2.3168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5119 -2.3168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1623 -1.7112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1623 -1.7112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4631 -1.7112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4631 -1.7112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1135 -2.3168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1135 -2.3168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4631 -2.9223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4631 -2.9223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1623 -2.9223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1623 -2.9223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4150 -2.3168 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4150 -2.3168 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5412 -0.4959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5412 -0.4959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1700 -0.9858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1700 -0.9858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6355 -0.7780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6355 -0.7780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1198 -0.7724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1198 -0.7724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4945 -0.3975 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4945 -0.3975 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9622 -0.6443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9622 -0.6443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0550 -0.7925 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0550 -0.7925 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7486 -1.0140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7486 -1.0140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3293 -1.2921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3293 -1.2921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5817 -0.0248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5817 -0.0248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5817 0.5598 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5817 0.5598 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0221 1.0002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0221 1.0002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5551 0.6924 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5551 0.6924 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0221 1.6121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0221 1.6121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5293 1.9049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5293 1.9049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9637 1.6541 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9637 1.6541 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5293 2.4588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5293 2.4588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6259 -1.3634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6259 -1.3634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2603 -0.9972 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2603 -0.9972 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6259 -2.1246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6259 -2.1246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1982 -2.4550 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1982 -2.4550 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8004 -2.1073 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8004 -2.1073 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1982 -3.0533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1982 -3.0533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1139 -3.5272 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1139 -3.5272 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 13 22 1 0 0 0 0 | + | 13 22 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 28 30 1 0 0 0 0 | + | 28 30 1 0 0 0 0 |
| − | 27 31 1 0 0 0 0 | + | 27 31 1 0 0 0 0 |
| − | 26 32 1 0 0 0 0 | + | 26 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 34 36 1 0 0 0 0 | + | 34 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 2 0 0 0 0 | + | 40 41 2 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 42 43 2 0 0 0 0 | + | 42 43 2 0 0 0 0 |
| − | 43 38 1 0 0 0 0 | + | 43 38 1 0 0 0 0 |
| − | 41 44 1 0 0 0 0 | + | 41 44 1 0 0 0 0 |
| − | 20 24 1 0 0 0 0 | + | 20 24 1 0 0 0 0 |
| − | 45 46 1 1 0 0 0 | + | 45 46 1 1 0 0 0 |
| − | 46 47 1 1 0 0 0 | + | 46 47 1 1 0 0 0 |
| − | 48 47 1 1 0 0 0 | + | 48 47 1 1 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 45 1 0 0 0 0 | + | 50 45 1 0 0 0 0 |
| − | 45 51 1 0 0 0 0 | + | 45 51 1 0 0 0 0 |
| − | 46 52 1 0 0 0 0 | + | 46 52 1 0 0 0 0 |
| − | 47 53 1 0 0 0 0 | + | 47 53 1 0 0 0 0 |
| − | 48 19 1 0 0 0 0 | + | 48 19 1 0 0 0 0 |
| − | 50 54 1 0 0 0 0 | + | 50 54 1 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 56 57 2 0 0 0 0 | + | 56 57 2 0 0 0 0 |
| − | 56 58 1 0 0 0 0 | + | 56 58 1 0 0 0 0 |
| − | 58 59 1 0 0 0 0 | + | 58 59 1 0 0 0 0 |
| − | 59 60 1 0 0 0 0 | + | 59 60 1 0 0 0 0 |
| − | 59 61 2 0 0 0 0 | + | 59 61 2 0 0 0 0 |
| − | 51 62 1 0 0 0 0 | + | 51 62 1 0 0 0 0 |
| − | 62 63 2 0 0 0 0 | + | 62 63 2 0 0 0 0 |
| − | 62 64 1 0 0 0 0 | + | 62 64 1 0 0 0 0 |
| − | 64 65 1 0 0 0 0 | + | 64 65 1 0 0 0 0 |
| − | 65 66 1 0 0 0 0 | + | 65 66 1 0 0 0 0 |
| − | 65 67 2 0 0 0 0 | + | 65 67 2 0 0 0 0 |
| − | 42 68 1 0 0 0 0 | + | 42 68 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AAGGL0026 | + | ID FL7AAGGL0026 |
| − | KNApSAcK_ID C00006889 | + | KNApSAcK_ID C00006889 |
| − | NAME Salviadelphin | + | NAME Salviadelphin |
| − | CAS_RN 128508-45-2 | + | CAS_RN 128508-45-2 |
| − | FORMULA C42H41O26 | + | FORMULA C42H41O26 |
| − | EXACTMASS 961.188606484 | + | EXACTMASS 961.188606484 |
| − | AVERAGEMASS 961.7593400000001 | + | AVERAGEMASS 961.7593400000001 |
| − | SMILES c(c6)(cc(c(O)c6O)O)c([o+1]5)c(cc(c54)c(cc(O)c4)OC(C(O)3)OC(C(OC(CC(O)=O)=O)C(O)3)COC(=O)CC(O)=O)OC(C1O)OC(COC(=O)C=Cc(c2)cc(O)c(c2)O)C(C1O)O | + | SMILES c(c6)(cc(c(O)c6O)O)c([o+1]5)c(cc(c54)c(cc(O)c4)OC(C(O)3)OC(C(OC(CC(O)=O)=O)C(O)3)COC(=O)CC(O)=O)OC(C1O)OC(COC(=O)C=Cc(c2)cc(O)c(c2)O)C(C1O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
68 73 0 0 0 0 0 0 0 0999 V2000
-1.3512 1.5696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3512 0.9273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7949 0.6061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2386 0.9273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2386 1.5696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7949 1.8908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3177 0.6061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8740 0.9273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8740 1.5696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3177 1.8908 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
1.4301 1.8907 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9970 1.5633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5640 1.8907 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5640 2.5454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9970 2.8727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4301 2.5454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9073 1.8907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1308 2.8726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7949 -0.0360 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2901 0.2066 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9970 3.5272 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1300 1.5639 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9425 0.3004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6417 -0.2206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2202 -0.0553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8021 -0.2206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1030 0.3004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5245 0.1351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3838 0.1507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7627 0.5477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5371 0.0498 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3537 -0.2206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8004 -0.9943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4184 -1.6559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6988 -2.1416 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6552 -1.6559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2737 -2.3168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5119 -2.3168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1623 -1.7112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4631 -1.7112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1135 -2.3168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4631 -2.9223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1623 -2.9223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4150 -2.3168 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5412 -0.4959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1700 -0.9858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6355 -0.7780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1198 -0.7724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4945 -0.3975 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9622 -0.6443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0550 -0.7925 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7486 -1.0140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3293 -1.2921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5817 -0.0248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5817 0.5598 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0221 1.0002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5551 0.6924 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0221 1.6121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5293 1.9049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9637 1.6541 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5293 2.4588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6259 -1.3634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2603 -0.9972 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6259 -2.1246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1982 -2.4550 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8004 -2.1073 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1982 -3.0533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1139 -3.5272 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
15 21 1 0 0 0 0
13 22 1 0 0 0 0
23 24 1 0 0 0 0
24 25 1 1 0 0 0
25 26 1 1 0 0 0
27 26 1 1 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
23 29 1 0 0 0 0
28 30 1 0 0 0 0
27 31 1 0 0 0 0
26 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 35 2 0 0 0 0
34 36 1 0 0 0 0
36 37 2 0 0 0 0
37 38 1 0 0 0 0
38 39 2 0 0 0 0
39 40 1 0 0 0 0
40 41 2 0 0 0 0
41 42 1 0 0 0 0
42 43 2 0 0 0 0
43 38 1 0 0 0 0
41 44 1 0 0 0 0
20 24 1 0 0 0 0
45 46 1 1 0 0 0
46 47 1 1 0 0 0
48 47 1 1 0 0 0
48 49 1 0 0 0 0
49 50 1 0 0 0 0
50 45 1 0 0 0 0
45 51 1 0 0 0 0
46 52 1 0 0 0 0
47 53 1 0 0 0 0
48 19 1 0 0 0 0
50 54 1 0 0 0 0
54 55 1 0 0 0 0
55 56 1 0 0 0 0
56 57 2 0 0 0 0
56 58 1 0 0 0 0
58 59 1 0 0 0 0
59 60 1 0 0 0 0
59 61 2 0 0 0 0
51 62 1 0 0 0 0
62 63 2 0 0 0 0
62 64 1 0 0 0 0
64 65 1 0 0 0 0
65 66 1 0 0 0 0
65 67 2 0 0 0 0
42 68 1 0 0 0 0
S SKP 8
ID FL7AAGGL0026
KNApSAcK_ID C00006889
NAME Salviadelphin
CAS_RN 128508-45-2
FORMULA C42H41O26
EXACTMASS 961.188606484
AVERAGEMASS 961.7593400000001
SMILES c(c6)(cc(c(O)c6O)O)c([o+1]5)c(cc(c54)c(cc(O)c4)OC(C(O)3)OC(C(OC(CC(O)=O)=O)C(O)3)COC(=O)CC(O)=O)OC(C1O)OC(COC(=O)C=Cc(c2)cc(O)c(c2)O)C(C1O)O
M END