Mol:FL7AAGGL0032
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 88 96 0 0 0 0 0 0 0 0999 V2000 | + | 88 96 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.6907 -1.1278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6907 -1.1278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0897 -0.7808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0897 -0.7808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0897 -0.0868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0897 -0.0868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6907 0.2602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6907 0.2602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2917 -0.0868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2917 -0.0868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2917 -0.7808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2917 -0.7808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4887 -1.1278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4887 -1.1278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8877 -0.7808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8877 -0.7808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8877 -0.0868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8877 -0.0868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4887 0.2602 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -2.4887 0.2602 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2519 0.2803 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2519 0.2803 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6834 -0.0479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6834 -0.0479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1149 0.2803 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1149 0.2803 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1149 0.9367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1149 0.9367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6834 1.2649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6834 1.2649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2519 0.9367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2519 0.9367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3992 1.2336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3992 1.2336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8527 0.2371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8527 0.2371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6907 -1.7548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6907 -1.7548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5615 -1.4064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5615 -1.4064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6834 1.7928 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6834 1.7928 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4033 -0.0189 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4033 -0.0189 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.5304 0.1663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.5304 0.1663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.9594 -0.4292 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.9594 -0.4292 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.2343 -0.0358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.2343 -0.0358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4098 -0.0669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4098 -0.0669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.9808 0.5287 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.9808 0.5287 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.7060 0.1354 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.7060 0.1354 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.9691 0.6140 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.9691 0.6140 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.4258 0.7467 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.4258 0.7467 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.8067 -0.2899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.8067 -0.2899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.3961 -0.4200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.3961 -0.4200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.9688 -0.5187 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.9688 -0.5187 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5495 -1.3198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5495 -1.3198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0862 -1.9464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0862 -1.9464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2648 -1.8692 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2648 -1.8692 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4813 -2.2213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4813 -2.2213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0180 -1.5948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0180 -1.5948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1966 -1.6719 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1966 -1.6719 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9941 -1.4389 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9941 -1.4389 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6205 -1.2480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6205 -1.2480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3758 -1.9526 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3758 -1.9526 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0243 -2.0758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0243 -2.0758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4359 -2.4874 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4359 -2.4874 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3416 2.2301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3416 2.2301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0709 1.5157 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0709 1.5157 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7224 1.7424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7224 1.7424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5204 1.5332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5204 1.5332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9330 2.2477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9330 2.2477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1397 2.0210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1397 2.0210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2598 2.0690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2598 2.0690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4090 2.4874 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4090 2.4874 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3419 2.0116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3419 2.0116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0241 1.8240 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0241 1.8240 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6005 1.4912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6005 1.4912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5552 0.1745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5552 0.1745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9916 -0.4279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9916 -0.4279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8156 -0.3869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8156 -0.3869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5455 -0.7714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5455 -0.7714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1092 -0.1691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1092 -0.1691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2852 -0.2100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2852 -0.2100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9329 0.1538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9329 0.1538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6532 0.1832 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6532 0.1832 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4539 -0.4916 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4539 -0.4916 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1019 -0.6024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1019 -0.6024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5879 -1.0571 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5879 -1.0571 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2359 1.8580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2359 1.8580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2359 2.4780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2359 2.4780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8467 1.5053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8467 1.5053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4974 1.8810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4974 1.8810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1178 1.5229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1178 1.5229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1178 0.8571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1178 0.8571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6944 0.5242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6944 0.5242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.2710 0.8571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.2710 0.8571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.2710 1.5229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.2710 1.5229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6944 1.8558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6944 1.8558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.6820 0.6198 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.6820 0.6198 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3861 -0.8432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3861 -0.8432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3861 -0.2340 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3861 -0.2340 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0396 -1.2205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0396 -1.2205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6557 -0.8648 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6557 -0.8648 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.2375 -1.2007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.2375 -1.2007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.2375 -1.8539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.2375 -1.8539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8032 -2.1805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8032 -2.1805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.3689 -1.8539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.3689 -1.8539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.3689 -1.2007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.3689 -1.2007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8032 -0.8740 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8032 -0.8740 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.8067 -2.1066 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.8067 -2.1066 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 3 1 0 0 0 0 | + | 10 3 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 5 18 1 0 0 0 0 | + | 5 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 13 22 1 0 0 0 0 | + | 13 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 33 1 0 0 0 0 | + | 25 33 1 0 0 0 0 |
| − | 18 26 1 0 0 0 0 | + | 18 26 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 38 37 1 1 0 0 0 | + | 38 37 1 1 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 20 35 1 0 0 0 0 | + | 20 35 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 39 41 1 0 0 0 0 | + | 39 41 1 0 0 0 0 |
| − | 38 42 1 0 0 0 0 | + | 38 42 1 0 0 0 0 |
| − | 37 43 1 0 0 0 0 | + | 37 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 1 1 0 0 0 | + | 46 47 1 1 0 0 0 |
| − | 48 47 1 1 0 0 0 | + | 48 47 1 1 0 0 0 |
| − | 49 48 1 1 0 0 0 | + | 49 48 1 1 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 45 1 0 0 0 0 | + | 50 45 1 0 0 0 0 |
| − | 45 51 1 0 0 0 0 | + | 45 51 1 0 0 0 0 |
| − | 50 52 1 0 0 0 0 | + | 50 52 1 0 0 0 0 |
| − | 49 53 1 0 0 0 0 | + | 49 53 1 0 0 0 0 |
| − | 48 54 1 0 0 0 0 | + | 48 54 1 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 21 46 1 0 0 0 0 | + | 21 46 1 0 0 0 0 |
| − | 56 57 1 0 0 0 0 | + | 56 57 1 0 0 0 0 |
| − | 57 58 1 1 0 0 0 | + | 57 58 1 1 0 0 0 |
| − | 59 58 1 1 0 0 0 | + | 59 58 1 1 0 0 0 |
| − | 60 59 1 1 0 0 0 | + | 60 59 1 1 0 0 0 |
| − | 60 61 1 0 0 0 0 | + | 60 61 1 0 0 0 0 |
| − | 61 56 1 0 0 0 0 | + | 61 56 1 0 0 0 0 |
| − | 56 62 1 0 0 0 0 | + | 56 62 1 0 0 0 0 |
| − | 61 63 1 0 0 0 0 | + | 61 63 1 0 0 0 0 |
| − | 60 64 1 0 0 0 0 | + | 60 64 1 0 0 0 0 |
| − | 59 65 1 0 0 0 0 | + | 59 65 1 0 0 0 0 |
| − | 65 66 1 0 0 0 0 | + | 65 66 1 0 0 0 0 |
| − | 22 57 1 0 0 0 0 | + | 22 57 1 0 0 0 0 |
| − | 55 67 1 0 0 0 0 | + | 55 67 1 0 0 0 0 |
| − | 67 68 2 0 0 0 0 | + | 67 68 2 0 0 0 0 |
| − | 67 69 1 0 0 0 0 | + | 67 69 1 0 0 0 0 |
| − | 69 70 2 0 0 0 0 | + | 69 70 2 0 0 0 0 |
| − | 70 71 1 0 0 0 0 | + | 70 71 1 0 0 0 0 |
| − | 71 72 2 0 0 0 0 | + | 71 72 2 0 0 0 0 |
| − | 72 73 1 0 0 0 0 | + | 72 73 1 0 0 0 0 |
| − | 73 74 2 0 0 0 0 | + | 73 74 2 0 0 0 0 |
| − | 74 75 1 0 0 0 0 | + | 74 75 1 0 0 0 0 |
| − | 75 76 2 0 0 0 0 | + | 75 76 2 0 0 0 0 |
| − | 76 71 1 0 0 0 0 | + | 76 71 1 0 0 0 0 |
| − | 74 77 1 0 0 0 0 | + | 74 77 1 0 0 0 0 |
| − | 66 78 1 0 0 0 0 | + | 66 78 1 0 0 0 0 |
| − | 78 79 2 0 0 0 0 | + | 78 79 2 0 0 0 0 |
| − | 78 80 1 0 0 0 0 | + | 78 80 1 0 0 0 0 |
| − | 80 81 2 0 0 0 0 | + | 80 81 2 0 0 0 0 |
| − | 81 82 1 0 0 0 0 | + | 81 82 1 0 0 0 0 |
| − | 82 83 2 0 0 0 0 | + | 82 83 2 0 0 0 0 |
| − | 83 84 1 0 0 0 0 | + | 83 84 1 0 0 0 0 |
| − | 84 85 2 0 0 0 0 | + | 84 85 2 0 0 0 0 |
| − | 85 86 1 0 0 0 0 | + | 85 86 1 0 0 0 0 |
| − | 86 87 2 0 0 0 0 | + | 86 87 2 0 0 0 0 |
| − | 87 82 1 0 0 0 0 | + | 87 82 1 0 0 0 0 |
| − | 85 88 1 0 0 0 0 | + | 85 88 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AAGGL0032 | + | ID FL7AAGGL0032 |
| − | KNApSAcK_ID C00011103 | + | KNApSAcK_ID C00011103 |
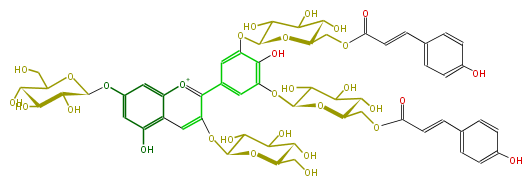
| − | NAME Delphinidin 3,7-diglucoside-3',5'-di(6-O-p-coumaroyl-beta-glucoside) | + | NAME Delphinidin 3,7-diglucoside-3',5'-di(6-O-p-coumaroyl-beta-glucoside) |
| − | CAS_RN 395680-63-4 | + | CAS_RN 395680-63-4 |
| − | FORMULA C57H63O31 | + | FORMULA C57H63O31 |
| − | EXACTMASS 1243.335330298 | + | EXACTMASS 1243.335330298 |
| − | AVERAGEMASS 1244.09152 | + | AVERAGEMASS 1244.09152 |
| − | SMILES OC(C8COC(=O)C=Cc(c9)ccc(c9)O)C(O)C(C(O8)Oc(c1O)cc(c(c6OC(C(O)7)OC(CO)C(O)C7O)[o+1]c(c(c6)5)cc(cc5O)OC(O4)C(C(C(O)C4CO)O)O)cc(OC(O2)C(C(O)C(O)C2COC(=O)C=Cc(c3)ccc(c3)O)O)1)O | + | SMILES OC(C8COC(=O)C=Cc(c9)ccc(c9)O)C(O)C(C(O8)Oc(c1O)cc(c(c6OC(C(O)7)OC(CO)C(O)C7O)[o+1]c(c(c6)5)cc(cc5O)OC(O4)C(C(C(O)C4CO)O)O)cc(OC(O2)C(C(O)C(O)C2COC(=O)C=Cc(c3)ccc(c3)O)O)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
88 96 0 0 0 0 0 0 0 0999 V2000
-3.6907 -1.1278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0897 -0.7808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0897 -0.0868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6907 0.2602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2917 -0.0868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2917 -0.7808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4887 -1.1278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8877 -0.7808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8877 -0.0868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4887 0.2602 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
-1.2519 0.2803 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6834 -0.0479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1149 0.2803 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1149 0.9367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6834 1.2649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2519 0.9367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3992 1.2336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8527 0.2371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6907 -1.7548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5615 -1.4064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6834 1.7928 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4033 -0.0189 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-7.5304 0.1663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.9594 -0.4292 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.2343 -0.0358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.4098 -0.0669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.9808 0.5287 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.7060 0.1354 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.9691 0.6140 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-7.4258 0.7467 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-7.8067 -0.2899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-7.3961 -0.4200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.9688 -0.5187 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5495 -1.3198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0862 -1.9464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2648 -1.8692 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4813 -2.2213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0180 -1.5948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1966 -1.6719 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9941 -1.4389 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6205 -1.2480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3758 -1.9526 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0243 -2.0758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4359 -2.4874 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3416 2.2301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0709 1.5157 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7224 1.7424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5204 1.5332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9330 2.2477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1397 2.0210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2598 2.0690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4090 2.4874 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3419 2.0116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0241 1.8240 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6005 1.4912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5552 0.1745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9916 -0.4279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8156 -0.3869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5455 -0.7714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1092 -0.1691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2852 -0.2100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9329 0.1538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6532 0.1832 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4539 -0.4916 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1019 -0.6024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5879 -1.0571 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2359 1.8580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2359 2.4780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8467 1.5053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4974 1.8810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1178 1.5229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1178 0.8571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.6944 0.5242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.2710 0.8571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.2710 1.5229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.6944 1.8558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.6820 0.6198 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3861 -0.8432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3861 -0.2340 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0396 -1.2205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.6557 -0.8648 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.2375 -1.2007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.2375 -1.8539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.8032 -2.1805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.3689 -1.8539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.3689 -1.2007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.8032 -0.8740 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.8067 -2.1066 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 3 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
14 17 1 0 0 0 0
5 18 1 0 0 0 0
1 19 1 0 0 0 0
8 20 1 0 0 0 0
15 21 1 0 0 0 0
13 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
28 29 1 0 0 0 0
29 30 1 0 0 0 0
23 31 1 0 0 0 0
24 32 1 0 0 0 0
25 33 1 0 0 0 0
18 26 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 1 0 0 0
36 37 1 1 0 0 0
38 37 1 1 0 0 0
38 39 1 0 0 0 0
39 34 1 0 0 0 0
20 35 1 0 0 0 0
34 40 1 0 0 0 0
39 41 1 0 0 0 0
38 42 1 0 0 0 0
37 43 1 0 0 0 0
43 44 1 0 0 0 0
45 46 1 0 0 0 0
46 47 1 1 0 0 0
48 47 1 1 0 0 0
49 48 1 1 0 0 0
49 50 1 0 0 0 0
50 45 1 0 0 0 0
45 51 1 0 0 0 0
50 52 1 0 0 0 0
49 53 1 0 0 0 0
48 54 1 0 0 0 0
54 55 1 0 0 0 0
21 46 1 0 0 0 0
56 57 1 0 0 0 0
57 58 1 1 0 0 0
59 58 1 1 0 0 0
60 59 1 1 0 0 0
60 61 1 0 0 0 0
61 56 1 0 0 0 0
56 62 1 0 0 0 0
61 63 1 0 0 0 0
60 64 1 0 0 0 0
59 65 1 0 0 0 0
65 66 1 0 0 0 0
22 57 1 0 0 0 0
55 67 1 0 0 0 0
67 68 2 0 0 0 0
67 69 1 0 0 0 0
69 70 2 0 0 0 0
70 71 1 0 0 0 0
71 72 2 0 0 0 0
72 73 1 0 0 0 0
73 74 2 0 0 0 0
74 75 1 0 0 0 0
75 76 2 0 0 0 0
76 71 1 0 0 0 0
74 77 1 0 0 0 0
66 78 1 0 0 0 0
78 79 2 0 0 0 0
78 80 1 0 0 0 0
80 81 2 0 0 0 0
81 82 1 0 0 0 0
82 83 2 0 0 0 0
83 84 1 0 0 0 0
84 85 2 0 0 0 0
85 86 1 0 0 0 0
86 87 2 0 0 0 0
87 82 1 0 0 0 0
85 88 1 0 0 0 0
S SKP 8
ID FL7AAGGL0032
KNApSAcK_ID C00011103
NAME Delphinidin 3,7-diglucoside-3',5'-di(6-O-p-coumaroyl-beta-glucoside)
CAS_RN 395680-63-4
FORMULA C57H63O31
EXACTMASS 1243.335330298
AVERAGEMASS 1244.09152
SMILES OC(C8COC(=O)C=Cc(c9)ccc(c9)O)C(O)C(C(O8)Oc(c1O)cc(c(c6OC(C(O)7)OC(CO)C(O)C7O)[o+1]c(c(c6)5)cc(cc5O)OC(O4)C(C(C(O)C4CO)O)O)cc(OC(O2)C(C(O)C(O)C2COC(=O)C=Cc(c3)ccc(c3)O)O)1)O
M END