Mol:FL7AAGGL0039
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 67 73 0 0 0 0 0 0 0 0999 V2000 | + | 67 73 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.0573 0.8233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0573 0.8233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0573 -0.0017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0573 -0.0017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7717 -0.4142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7717 -0.4142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4862 -0.0017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4862 -0.0017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4862 0.8233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4862 0.8233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7717 1.2358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7717 1.2358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2007 -0.4142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2007 -0.4142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9151 -0.0017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9151 -0.0017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9151 0.8233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9151 0.8233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2007 1.2358 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 2.2007 1.2358 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6917 1.2716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6917 1.2716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4061 0.8591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4061 0.8591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1206 1.2716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1206 1.2716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1206 2.0966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1206 2.0966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4061 2.5091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4061 2.5091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6917 2.0966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6917 2.0966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7608 2.4662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7608 2.4662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7736 -0.4974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7736 -0.4974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5227 1.1581 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5227 1.1581 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7717 -1.1769 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7717 -1.1769 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7655 0.8992 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7655 0.8992 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4061 3.2403 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4061 3.2403 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3069 -2.5247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3069 -2.5247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0654 -2.8499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0654 -2.8499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5795 -2.2043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5795 -2.2043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3508 -1.9108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3508 -1.9108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5923 -1.5854 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5923 -1.5854 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0782 -2.2310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0782 -2.2310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2213 -2.4795 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2213 -2.4795 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4496 -2.9232 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4496 -2.9232 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0001 -3.2403 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0001 -3.2403 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6677 -1.9079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6677 -1.9079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2237 -1.9608 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2237 -1.9608 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0270 -1.3058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0270 -1.3058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8493 -1.2361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8493 -1.2361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9994 -0.4245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9994 -0.4245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.5420 0.1972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5420 0.1972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7197 0.1276 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7197 0.1276 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5695 -0.6839 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5695 -0.6839 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.6953 -0.3656 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.6953 -0.3656 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3404 -1.5904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3404 -1.5904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0928 -2.0816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0928 -2.0816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0554 -0.5918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0554 -0.5918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6884 -0.8473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6884 -0.8473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7403 -1.0729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7403 -1.0729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3277 -1.7876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3277 -1.7876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5293 -1.5784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5293 -1.5784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2642 -1.8053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2642 -1.8053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1484 -1.0905 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1484 -1.0905 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9468 -1.2996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9468 -1.2996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1038 -0.7138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1038 -0.7138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2235 -1.5561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2235 -1.5561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7763 -1.5285 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7763 -1.5285 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7008 -2.2184 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7008 -2.2184 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7497 -0.4122 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7497 -0.4122 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1617 0.3013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1617 0.3013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7497 1.0148 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7497 1.0148 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9856 0.3013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9856 0.3013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3975 -0.4122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3975 -0.4122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2214 -0.4122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2214 -0.4122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6339 0.3023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6339 0.3023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4589 0.3023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4589 0.3023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8714 -0.4122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8714 -0.4122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4589 -1.1267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4589 -1.1267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6339 -1.1267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6339 -1.1267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.6953 -0.4122 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.6953 -0.4122 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8708 1.0158 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8708 1.0158 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 8 18 1 0 0 0 0 | + | 8 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 13 21 1 0 0 0 0 | + | 13 21 1 0 0 0 0 |
| − | 15 22 1 0 0 0 0 | + | 15 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 28 32 1 0 0 0 0 | + | 28 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 26 18 1 0 0 0 0 | + | 26 18 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 37 36 1 1 0 0 0 | + | 37 36 1 1 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 36 40 1 0 0 0 0 | + | 36 40 1 0 0 0 0 |
| − | 35 41 1 0 0 0 0 | + | 35 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 39 43 1 0 0 0 0 | + | 39 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 37 21 1 0 0 0 0 | + | 37 21 1 0 0 0 0 |
| − | 45 46 1 1 0 0 0 | + | 45 46 1 1 0 0 0 |
| − | 46 47 1 1 0 0 0 | + | 46 47 1 1 0 0 0 |
| − | 48 47 1 1 0 0 0 | + | 48 47 1 1 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 45 1 0 0 0 0 | + | 50 45 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 45 52 1 0 0 0 0 | + | 45 52 1 0 0 0 0 |
| − | 46 53 1 0 0 0 0 | + | 46 53 1 0 0 0 0 |
| − | 47 54 1 0 0 0 0 | + | 47 54 1 0 0 0 0 |
| − | 20 48 1 0 0 0 0 | + | 20 48 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 56 57 2 0 0 0 0 | + | 56 57 2 0 0 0 0 |
| − | 56 58 1 0 0 0 0 | + | 56 58 1 0 0 0 0 |
| − | 58 59 2 0 0 0 0 | + | 58 59 2 0 0 0 0 |
| − | 59 60 1 0 0 0 0 | + | 59 60 1 0 0 0 0 |
| − | 60 61 2 0 0 0 0 | + | 60 61 2 0 0 0 0 |
| − | 61 62 1 0 0 0 0 | + | 61 62 1 0 0 0 0 |
| − | 62 63 2 0 0 0 0 | + | 62 63 2 0 0 0 0 |
| − | 63 64 1 0 0 0 0 | + | 63 64 1 0 0 0 0 |
| − | 64 65 2 0 0 0 0 | + | 64 65 2 0 0 0 0 |
| − | 65 60 1 0 0 0 0 | + | 65 60 1 0 0 0 0 |
| − | 63 66 1 0 0 0 0 | + | 63 66 1 0 0 0 0 |
| − | 62 67 1 0 0 0 0 | + | 62 67 1 0 0 0 0 |
| − | 55 51 1 0 0 0 0 | + | 55 51 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AAGGL0039 | + | ID FL7AAGGL0039 |
| − | KNApSAcK_ID C00014781 | + | KNApSAcK_ID C00014781 |
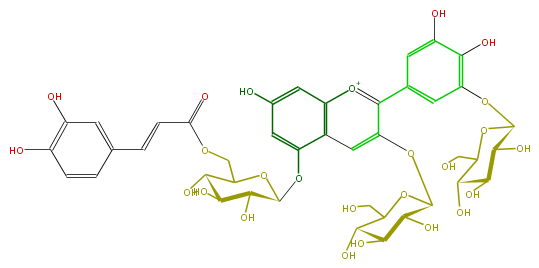
| − | NAME Delphinidin 3,3'-di-glucoside-5-(6-caffeoylglucoside) | + | NAME Delphinidin 3,3'-di-glucoside-5-(6-caffeoylglucoside) |
| − | CAS_RN 198645-07-7 | + | CAS_RN 198645-07-7 |
| − | FORMULA C42H47O25 | + | FORMULA C42H47O25 |
| − | EXACTMASS 951.2406420539999 | + | EXACTMASS 951.2406420539999 |
| − | AVERAGEMASS 951.80758 | + | AVERAGEMASS 951.80758 |
| − | SMILES c(c35)(cc(cc3[o+1]c(c(c6)cc(O)c(c6OC(C7O)OC(CO)C(C7O)O)O)c(c5)OC(C4O)OC(C(O)C4O)CO)O)OC(C(O)2)OC(C(C2O)O)COC(=O)C=Cc(c1)ccc(O)c1O | + | SMILES c(c35)(cc(cc3[o+1]c(c(c6)cc(O)c(c6OC(C7O)OC(CO)C(C7O)O)O)c(c5)OC(C4O)OC(C(O)C4O)CO)O)OC(C(O)2)OC(C(C2O)O)COC(=O)C=Cc(c1)ccc(O)c1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
67 73 0 0 0 0 0 0 0 0999 V2000
0.0573 0.8233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0573 -0.0017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7717 -0.4142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4862 -0.0017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4862 0.8233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7717 1.2358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2007 -0.4142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9151 -0.0017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9151 0.8233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2007 1.2358 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
3.6917 1.2716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4061 0.8591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1206 1.2716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1206 2.0966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4061 2.5091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6917 2.0966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.7608 2.4662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7736 -0.4974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5227 1.1581 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7717 -1.1769 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.7655 0.8992 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4061 3.2403 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3069 -2.5247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0654 -2.8499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5795 -2.2043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3508 -1.9108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5923 -1.5854 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0782 -2.2310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2213 -2.4795 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4496 -2.9232 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0001 -3.2403 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6677 -1.9079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2237 -1.9608 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0270 -1.3058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.8493 -1.2361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9994 -0.4245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.5420 0.1972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.7197 0.1276 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.5695 -0.6839 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.6953 -0.3656 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.3404 -1.5904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0928 -2.0816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0554 -0.5918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6884 -0.8473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7403 -1.0729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3277 -1.7876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5293 -1.5784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2642 -1.8053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1484 -1.0905 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9468 -1.2996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1038 -0.7138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2235 -1.5561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7763 -1.5285 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7008 -2.2184 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7497 -0.4122 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1617 0.3013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7497 1.0148 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9856 0.3013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3975 -0.4122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2214 -0.4122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6339 0.3023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.4589 0.3023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.8714 -0.4122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.4589 -1.1267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6339 -1.1267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.6953 -0.4122 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.8708 1.0158 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
14 17 1 0 0 0 0
8 18 1 0 0 0 0
1 19 1 0 0 0 0
3 20 1 0 0 0 0
13 21 1 0 0 0 0
15 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
25 29 1 0 0 0 0
24 30 1 0 0 0 0
23 31 1 0 0 0 0
28 32 1 0 0 0 0
32 33 1 0 0 0 0
26 18 1 0 0 0 0
34 35 1 1 0 0 0
35 36 1 1 0 0 0
37 36 1 1 0 0 0
37 38 1 0 0 0 0
38 39 1 0 0 0 0
39 34 1 0 0 0 0
36 40 1 0 0 0 0
35 41 1 0 0 0 0
34 42 1 0 0 0 0
39 43 1 0 0 0 0
43 44 1 0 0 0 0
37 21 1 0 0 0 0
45 46 1 1 0 0 0
46 47 1 1 0 0 0
48 47 1 1 0 0 0
48 49 1 0 0 0 0
49 50 1 0 0 0 0
50 45 1 0 0 0 0
50 51 1 0 0 0 0
45 52 1 0 0 0 0
46 53 1 0 0 0 0
47 54 1 0 0 0 0
20 48 1 0 0 0 0
55 56 1 0 0 0 0
56 57 2 0 0 0 0
56 58 1 0 0 0 0
58 59 2 0 0 0 0
59 60 1 0 0 0 0
60 61 2 0 0 0 0
61 62 1 0 0 0 0
62 63 2 0 0 0 0
63 64 1 0 0 0 0
64 65 2 0 0 0 0
65 60 1 0 0 0 0
63 66 1 0 0 0 0
62 67 1 0 0 0 0
55 51 1 0 0 0 0
S SKP 8
ID FL7AAGGL0039
KNApSAcK_ID C00014781
NAME Delphinidin 3,3'-di-glucoside-5-(6-caffeoylglucoside)
CAS_RN 198645-07-7
FORMULA C42H47O25
EXACTMASS 951.2406420539999
AVERAGEMASS 951.80758
SMILES c(c35)(cc(cc3[o+1]c(c(c6)cc(O)c(c6OC(C7O)OC(CO)C(C7O)O)O)c(c5)OC(C4O)OC(C(O)C4O)CO)O)OC(C(O)2)OC(C(C2O)O)COC(=O)C=Cc(c1)ccc(O)c1O
M END