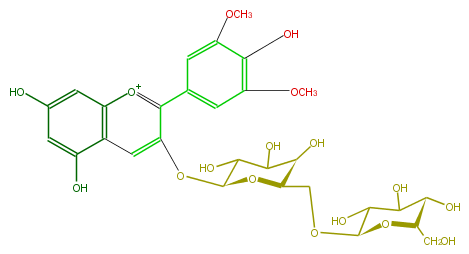
Mol:FL7AAIGL0004
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 46 50 0 0 0 0 0 0 0 0999 V2000 | + | 46 50 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.3092 -1.8754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3092 -1.8754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4667 -1.5753 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.4667 -1.5753 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.1659 -2.0963 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.1659 -2.0963 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.7443 -1.9310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7443 -1.9310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3263 -2.0963 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.3263 -2.0963 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.6272 -1.5753 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.6272 -1.5753 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.0487 -1.7406 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 3.0487 -1.7406 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.9080 -1.7250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9080 -1.7250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0902 -1.2660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0902 -1.2660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0612 -1.8259 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0612 -1.8259 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4941 1.5233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4941 1.5233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6056 0.8907 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6056 0.8907 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1136 0.4779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1136 0.4779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5099 0.6975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5099 0.6975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3984 1.3301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3984 1.3301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8905 1.7430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8905 1.7430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0179 0.2847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0179 0.2847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4142 0.5043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4142 0.5043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3027 1.1369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3027 1.1369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7948 1.5498 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -1.7948 1.5498 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6993 1.3566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6993 1.3566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1978 0.9357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1978 0.9357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4174 1.1597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4174 1.1597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5311 1.8043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5311 1.8043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0296 2.2252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0296 2.2252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5856 2.0012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5856 2.0012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9860 1.9361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9860 1.9361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4708 2.1463 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4708 2.1463 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2250 -0.1545 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2250 -0.1545 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1296 -0.2777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1296 -0.2777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0623 -0.1832 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.0623 -0.1832 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.3243 -0.6439 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.3243 -0.6439 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.2740 -0.5817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2740 -0.5817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8186 -0.8455 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.8186 -0.8455 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.2052 -0.3847 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.2052 -0.3847 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.6068 -0.4470 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.6068 -0.4470 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.5139 -0.2336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5139 -0.2336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7301 0.0132 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7301 0.0132 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6158 -0.0972 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6158 -0.0972 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3616 -0.9413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3616 -0.9413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4968 -2.4177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4968 -2.4177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4968 -3.2428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4968 -3.2428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4022 0.9860 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4022 0.9860 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3870 0.8124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3870 0.8124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4088 2.7433 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4088 2.7433 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9993 3.5504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9993 3.5504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 1 1 0 0 0 | + | 3 4 1 1 0 0 0 |
| − | 4 5 1 1 0 0 0 | + | 4 5 1 1 0 0 0 |
| − | 6 5 1 1 0 0 0 | + | 6 5 1 1 0 0 0 |
| − | 6 7 1 0 0 0 0 | + | 6 7 1 0 0 0 0 |
| − | 7 2 1 0 0 0 0 | + | 7 2 1 0 0 0 0 |
| − | 2 8 1 0 0 0 0 | + | 2 8 1 0 0 0 0 |
| − | 7 9 1 0 0 0 0 | + | 7 9 1 0 0 0 0 |
| − | 6 10 1 0 0 0 0 | + | 6 10 1 0 0 0 0 |
| − | 3 1 1 0 0 0 0 | + | 3 1 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 11 2 0 0 0 0 | + | 16 11 2 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 19 20 2 0 0 0 0 | + | 19 20 2 0 0 0 0 |
| − | 20 15 1 0 0 0 0 | + | 20 15 1 0 0 0 0 |
| − | 19 21 1 0 0 0 0 | + | 19 21 1 0 0 0 0 |
| − | 21 22 2 0 0 0 0 | + | 21 22 2 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 2 0 0 0 0 | + | 23 24 2 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 2 0 0 0 0 | + | 25 26 2 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 11 27 1 0 0 0 0 | + | 11 27 1 0 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 13 29 1 0 0 0 0 | + | 13 29 1 0 0 0 0 |
| − | 30 18 1 0 0 0 0 | + | 30 18 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 32 30 1 0 0 0 0 | + | 32 30 1 0 0 0 0 |
| − | 40 1 1 0 0 0 0 | + | 40 1 1 0 0 0 0 |
| − | 5 41 1 0 0 0 0 | + | 5 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 23 43 1 0 0 0 0 | + | 23 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 25 45 1 0 0 0 0 | + | 25 45 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 41 42 | + | M SAL 3 2 41 42 |
| − | M SBL 3 1 45 | + | M SBL 3 1 45 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SVB 3 45 3.6899 -2.1094 | + | M SVB 3 45 3.6899 -2.1094 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 45 46 | + | M SAL 2 2 45 46 |
| − | M SBL 2 1 49 | + | M SBL 2 1 49 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 49 -0.2166 2.5219 | + | M SVB 2 49 -0.2166 2.5219 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 43 44 | + | M SAL 1 2 43 44 |
| − | M SBL 1 1 47 | + | M SBL 1 1 47 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 47 0.2765 0.7575 | + | M SVB 1 47 0.2765 0.7575 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AAIGL0004 | + | ID FL7AAIGL0004 |
| − | KNApSAcK_ID C00006739 | + | KNApSAcK_ID C00006739 |
| − | NAME Malvidin 3-gentiobiside | + | NAME Malvidin 3-gentiobiside |
| − | CAS_RN - | + | CAS_RN - |
| − | FORMULA C29H35O17 | + | FORMULA C29H35O17 |
| − | EXACTMASS 655.187424694 | + | EXACTMASS 655.187424694 |
| − | AVERAGEMASS 655.578 | + | AVERAGEMASS 655.578 |
| − | SMILES C([C@@H](O)1)(C(O)[C@H](OC[C@H]([C@@H]5O)O[C@H](C(C5O)O)Oc(c3)c(c(c4)cc(OC)c(c(OC)4)O)[o+1]c(c32)cc(O)cc2O)O[C@H](CO)1)O | + | SMILES C([C@@H](O)1)(C(O)[C@H](OC[C@H]([C@@H]5O)O[C@H](C(C5O)O)Oc(c3)c(c(c4)cc(OC)c(c(OC)4)O)[o+1]c(c32)cc(O)cc2O)O[C@H](CO)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
46 50 0 0 0 0 0 0 0 0999 V2000
1.3092 -1.8754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4667 -1.5753 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.1659 -2.0963 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.7443 -1.9310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3263 -2.0963 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.6272 -1.5753 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.0487 -1.7406 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.9080 -1.7250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0902 -1.2660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0612 -1.8259 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4941 1.5233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6056 0.8907 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1136 0.4779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5099 0.6975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3984 1.3301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8905 1.7430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0179 0.2847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4142 0.5043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3027 1.1369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7948 1.5498 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
-0.6993 1.3566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1978 0.9357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4174 1.1597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5311 1.8043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0296 2.2252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5856 2.0012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9860 1.9361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4708 2.1463 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2250 -0.1545 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1296 -0.2777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0623 -0.1832 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.3243 -0.6439 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.2740 -0.5817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8186 -0.8455 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.2052 -0.3847 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.6068 -0.4470 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.5139 -0.2336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7301 0.0132 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6158 -0.0972 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3616 -0.9413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4968 -2.4177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4968 -3.2428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4022 0.9860 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3870 0.8124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4088 2.7433 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9993 3.5504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2 3 1 0 0 0 0
3 4 1 1 0 0 0
4 5 1 1 0 0 0
6 5 1 1 0 0 0
6 7 1 0 0 0 0
7 2 1 0 0 0 0
2 8 1 0 0 0 0
7 9 1 0 0 0 0
6 10 1 0 0 0 0
3 1 1 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 11 2 0 0 0 0
14 17 1 0 0 0 0
17 18 2 0 0 0 0
18 19 1 0 0 0 0
19 20 2 0 0 0 0
20 15 1 0 0 0 0
19 21 1 0 0 0 0
21 22 2 0 0 0 0
22 23 1 0 0 0 0
23 24 2 0 0 0 0
24 25 1 0 0 0 0
25 26 2 0 0 0 0
26 21 1 0 0 0 0
11 27 1 0 0 0 0
24 28 1 0 0 0 0
13 29 1 0 0 0 0
30 18 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
36 38 1 0 0 0 0
35 39 1 0 0 0 0
34 40 1 0 0 0 0
32 30 1 0 0 0 0
40 1 1 0 0 0 0
5 41 1 0 0 0 0
41 42 1 0 0 0 0
23 43 1 0 0 0 0
43 44 1 0 0 0 0
25 45 1 0 0 0 0
45 46 1 0 0 0 0
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 41 42
M SBL 3 1 45
M SMT 3 CH2OH
M SVB 3 45 3.6899 -2.1094
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 45 46
M SBL 2 1 49
M SMT 2 OCH3
M SVB 2 49 -0.2166 2.5219
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 43 44
M SBL 1 1 47
M SMT 1 OCH3
M SVB 1 47 0.2765 0.7575
S SKP 8
ID FL7AAIGL0004
KNApSAcK_ID C00006739
NAME Malvidin 3-gentiobiside
CAS_RN -
FORMULA C29H35O17
EXACTMASS 655.187424694
AVERAGEMASS 655.578
SMILES C([C@@H](O)1)(C(O)[C@H](OC[C@H]([C@@H]5O)O[C@H](C(C5O)O)Oc(c3)c(c(c4)cc(OC)c(c(OC)4)O)[o+1]c(c32)cc(O)cc2O)O[C@H](CO)1)O
M END