Mol:FL7AAIGO0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 46 50 0 0 0 0 0 0 0 0999 V2000 | + | 46 50 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.9513 -0.4345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9513 -0.4345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9513 -1.2119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9513 -1.2119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2779 -1.6007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2779 -1.6007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6047 -1.2119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6047 -1.2119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6047 -0.4345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6047 -0.4345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2779 -0.0457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2779 -0.0457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0686 -1.6007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0686 -1.6007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7418 -1.2119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7418 -1.2119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7418 -0.4345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7418 -0.4345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0686 -0.0457 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 0.0686 -0.0457 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4147 -0.0460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4147 -0.0460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0969 -0.4399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0969 -0.4399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7790 -0.0460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7790 -0.0460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7790 0.7417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7790 0.7417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0969 1.1355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0969 1.1355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4147 0.7417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4147 0.7417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4655 -1.6917 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4655 -1.6917 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6902 -1.5817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6902 -1.5817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0935 -2.1784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0935 -2.1784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9373 -2.1645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9373 -2.1645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6657 -2.5997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6657 -2.5997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2625 -2.0030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2625 -2.0030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4187 -2.0170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4187 -2.0170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9783 -1.4594 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9783 -1.4594 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1942 -1.5046 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1942 -1.5046 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9620 -2.3035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9620 -2.3035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6241 -0.0460 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6241 -0.0460 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2779 -2.3776 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2779 -2.3776 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4953 1.1553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4953 1.1553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0916 -1.9685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0916 -1.9685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6975 -2.6512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6975 -2.6512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9349 -2.4515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9349 -2.4515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1770 -2.6512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1770 -2.6512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5711 -1.9685 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5711 -1.9685 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3337 -2.1683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3337 -2.1683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3489 -3.1027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3489 -3.1027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8369 -2.1216 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8369 -2.1216 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4186 -2.5082 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4186 -2.5082 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9328 -1.4925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9328 -1.4925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9620 -1.1777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9620 -1.1777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3682 -2.6519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3682 -2.6519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9507 -2.8080 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9507 -2.8080 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0969 1.8772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0969 1.8772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6520 3.1027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6520 3.1027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4622 -0.4405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4622 -0.4405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8075 -0.4405 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8075 -0.4405 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 8 17 1 0 0 0 0 | + | 8 17 1 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 19 20 1 1 0 0 0 | + | 19 20 1 1 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 18 1 0 0 0 0 | + | 23 18 1 0 0 0 0 |
| − | 19 17 1 0 0 0 0 | + | 19 17 1 0 0 0 0 |
| − | 18 24 1 0 0 0 0 | + | 18 24 1 0 0 0 0 |
| − | 23 25 1 0 0 0 0 | + | 23 25 1 0 0 0 0 |
| − | 22 26 1 0 0 0 0 | + | 22 26 1 0 0 0 0 |
| − | 1 27 1 0 0 0 0 | + | 1 27 1 0 0 0 0 |
| − | 3 28 1 0 0 0 0 | + | 3 28 1 0 0 0 0 |
| − | 14 29 1 0 0 0 0 | + | 14 29 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 28 33 1 0 0 0 0 | + | 28 33 1 0 0 0 0 |
| − | 32 36 1 0 0 0 0 | + | 32 36 1 0 0 0 0 |
| − | 30 37 1 0 0 0 0 | + | 30 37 1 0 0 0 0 |
| − | 31 38 1 0 0 0 0 | + | 31 38 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 42 41 1 0 0 0 0 | + | 42 41 1 0 0 0 0 |
| − | 21 41 1 0 0 0 0 | + | 21 41 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 15 43 1 0 0 0 0 | + | 15 43 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 13 45 1 0 0 0 0 | + | 13 45 1 0 0 0 0 |
| − | M CHG 1 10 1 | + | M CHG 1 10 1 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 39 40 | + | M SAL 1 2 39 40 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 44 0.5991 -0.6758 | + | M SBV 1 44 0.5991 -0.6758 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 41 42 | + | M SAL 2 2 41 42 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 46 -0.7025 0.0522 | + | M SBV 2 46 -0.7025 0.0522 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 43 44 | + | M SAL 3 2 43 44 |
| − | M SBL 3 1 48 | + | M SBL 3 1 48 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SBV 3 48 0.0000 -0.7417 | + | M SBV 3 48 0.0000 -0.7417 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 45 46 | + | M SAL 4 2 45 46 |
| − | M SBL 4 1 50 | + | M SBL 4 1 50 |
| − | M SMT 4 OCH3 | + | M SMT 4 OCH3 |
| − | M SBV 4 50 -0.6832 0.3945 | + | M SBV 4 50 -0.6832 0.3945 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL7AAIGS0001 | + | ID FL7AAIGS0001 |
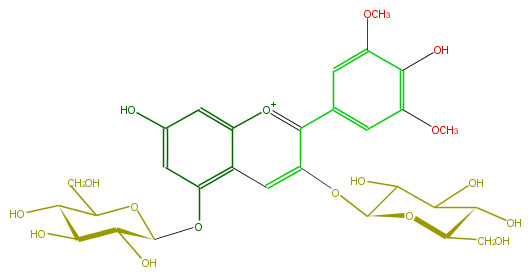
| − | FORMULA C29H35O17 | + | FORMULA C29H35O17 |
| − | EXACTMASS 655.187424694 | + | EXACTMASS 655.187424694 |
| − | AVERAGEMASS 655.578 | + | AVERAGEMASS 655.578 |
| − | SMILES Oc(c1)cc([o+1]4)c(cc(c4c(c5)cc(OC)c(c(OC)5)O)OC(O3)C(C(C(C(CO)3)O)O)O)c1OC(C(O)2)OC(C(C(O)2)O)CO | + | SMILES Oc(c1)cc([o+1]4)c(cc(c4c(c5)cc(OC)c(c(OC)5)O)OC(O3)C(C(C(C(CO)3)O)O)O)c1OC(C(O)2)OC(C(C(O)2)O)CO |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
46 50 0 0 0 0 0 0 0 0999 V2000
-1.9513 -0.4345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9513 -1.2119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2779 -1.6007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6047 -1.2119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6047 -0.4345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2779 -0.0457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0686 -1.6007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7418 -1.2119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7418 -0.4345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0686 -0.0457 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
1.4147 -0.0460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0969 -0.4399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7790 -0.0460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7790 0.7417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0969 1.1355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4147 0.7417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4655 -1.6917 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6902 -1.5817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0935 -2.1784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9373 -2.1645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6657 -2.5997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2625 -2.0030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4187 -2.0170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9783 -1.4594 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1942 -1.5046 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9620 -2.3035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6241 -0.0460 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2779 -2.3776 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4953 1.1553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0916 -1.9685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6975 -2.6512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9349 -2.4515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1770 -2.6512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5711 -1.9685 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3337 -2.1683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3489 -3.1027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8369 -2.1216 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4186 -2.5082 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9328 -1.4925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9620 -1.1777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3682 -2.6519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9507 -2.8080 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0969 1.8772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6520 3.1027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4622 -0.4405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8075 -0.4405 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
8 17 1 0 0 0 0
18 19 1 0 0 0 0
19 20 1 1 0 0 0
20 21 1 1 0 0 0
22 21 1 1 0 0 0
22 23 1 0 0 0 0
23 18 1 0 0 0 0
19 17 1 0 0 0 0
18 24 1 0 0 0 0
23 25 1 0 0 0 0
22 26 1 0 0 0 0
1 27 1 0 0 0 0
3 28 1 0 0 0 0
14 29 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
28 33 1 0 0 0 0
32 36 1 0 0 0 0
30 37 1 0 0 0 0
31 38 1 0 0 0 0
39 40 1 0 0 0 0
35 39 1 0 0 0 0
42 41 1 0 0 0 0
21 41 1 0 0 0 0
43 44 1 0 0 0 0
15 43 1 0 0 0 0
45 46 1 0 0 0 0
13 45 1 0 0 0 0
M CHG 1 10 1
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 39 40
M SBL 1 1 44
M SMT 1 ^ CH2OH
M SBV 1 44 0.5991 -0.6758
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 41 42
M SBL 2 1 46
M SMT 2 CH2OH
M SBV 2 46 -0.7025 0.0522
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 43 44
M SBL 3 1 48
M SMT 3 OCH3
M SBV 3 48 0.0000 -0.7417
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 45 46
M SBL 4 1 50
M SMT 4 OCH3
M SBV 4 50 -0.6832 0.3945
S SKP 5
ID FL7AAIGS0001
FORMULA C29H35O17
EXACTMASS 655.187424694
AVERAGEMASS 655.578
SMILES Oc(c1)cc([o+1]4)c(cc(c4c(c5)cc(OC)c(c(OC)5)O)OC(O3)C(C(C(C(CO)3)O)O)O)c1OC(C(O)2)OC(C(C(O)2)O)CO
M END