Mol:FLIA1AGS0006
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 41 45 0 0 0 0 0 0 0 0999 V2000 | + | 41 45 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.4507 0.2414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4507 0.2414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8944 -0.0798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8944 -0.0798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3381 0.2414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3381 0.2414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2180 -0.0797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2180 -0.0797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2180 -0.7457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2180 -0.7457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7948 -1.0786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7948 -1.0786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3715 -0.7457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3715 -0.7457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3715 -0.0797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3715 -0.0797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7948 0.2533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7948 0.2533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8942 -0.7218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8942 -0.7218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3381 -1.0429 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3381 -1.0429 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7948 -1.7441 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7948 -1.7441 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9478 -1.0784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9478 -1.0784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9478 -1.7150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9478 -1.7150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4992 -2.0334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4992 -2.0334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0505 -1.7150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0505 -1.7150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0505 -1.0784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0505 -1.0784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4992 -0.7601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4992 -0.7601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5559 0.1516 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -3.5559 0.1516 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -3.1500 -0.2956 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.1500 -0.2956 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.7141 -0.1261 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.7141 -0.1261 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.2935 -0.1216 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.2935 -0.1216 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.5991 0.1841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5991 0.1841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0444 0.0243 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -3.0444 0.0243 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -3.9933 -0.0076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9933 -0.0076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2643 -0.7216 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2643 -0.7216 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4643 -0.5454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4643 -0.5454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4216 0.5714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4216 0.5714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5845 0.9548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5845 0.9548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9103 1.9262 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9103 1.9262 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9545 1.4209 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.9545 1.4209 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.5713 1.7534 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -3.5713 1.7534 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -3.0677 1.8344 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.0677 1.8344 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.0234 2.3398 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.0234 2.3398 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.4066 2.0073 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -3.4066 2.0073 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -3.3717 2.4074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3717 2.4074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3117 3.0422 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3117 3.0422 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1614 2.2570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1614 2.2570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8964 2.3163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8964 2.3163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9165 -2.2150 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9165 -2.2150 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7826 -2.7149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7826 -2.7149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 7 1 0 0 0 0 | + | 6 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 4 1 0 0 0 0 | + | 9 4 1 0 0 0 0 |
| − | 2 10 1 0 0 0 0 | + | 2 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 5 1 0 0 0 0 | + | 11 5 1 0 0 0 0 |
| − | 6 12 2 0 0 0 0 | + | 6 12 2 0 0 0 0 |
| − | 7 13 1 0 0 0 0 | + | 7 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 19 20 1 1 0 0 0 | + | 19 20 1 1 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 29 28 1 0 0 0 0 | + | 29 28 1 0 0 0 0 |
| − | 22 1 1 0 0 0 0 | + | 22 1 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 34 37 1 0 0 0 0 | + | 34 37 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 32 39 1 0 0 0 0 | + | 32 39 1 0 0 0 0 |
| − | 31 29 1 0 0 0 0 | + | 31 29 1 0 0 0 0 |
| − | 16 40 1 0 0 0 0 | + | 16 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 44 3.2446 -1.9213 | + | M SVB 1 44 3.2446 -1.9213 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FLIA1AGS0006 | + | ID FLIA1AGS0006 |
| − | KNApSAcK_ID C00010081 | + | KNApSAcK_ID C00010081 |
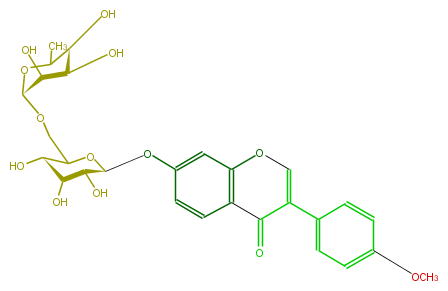
| − | NAME Formononetin 7-O-rutinoside | + | NAME Formononetin 7-O-rutinoside |
| − | CAS_RN 51351-35-0 | + | CAS_RN 51351-35-0 |
| − | FORMULA C28H32O13 | + | FORMULA C28H32O13 |
| − | EXACTMASS 576.18429111 | + | EXACTMASS 576.18429111 |
| − | AVERAGEMASS 576.54588 | + | AVERAGEMASS 576.54588 |
| − | SMILES O(C(C)1)[C@H](OCC([C@@H]2O)O[C@@H](Oc(c5)ccc(c53)C(C(c(c4)ccc(c4)OC)=CO3)=O)[C@H]([C@H]2O)O)[C@H]([C@@H]([C@H](O)1)O)O | + | SMILES O(C(C)1)[C@H](OCC([C@@H]2O)O[C@@H](Oc(c5)ccc(c53)C(C(c(c4)ccc(c4)OC)=CO3)=O)[C@H]([C@H]2O)O)[C@H]([C@@H]([C@H](O)1)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
41 45 0 0 0 0 0 0 0 0999 V2000
-1.4507 0.2414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8944 -0.0798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3381 0.2414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2180 -0.0797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2180 -0.7457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7948 -1.0786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3715 -0.7457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3715 -0.0797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7948 0.2533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8942 -0.7218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3381 -1.0429 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7948 -1.7441 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9478 -1.0784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9478 -1.7150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4992 -2.0334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0505 -1.7150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0505 -1.0784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4992 -0.7601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5559 0.1516 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-3.1500 -0.2956 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.7141 -0.1261 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.2935 -0.1216 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.5991 0.1841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0444 0.0243 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-3.9933 -0.0076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2643 -0.7216 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4643 -0.5454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4216 0.5714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5845 0.9548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9103 1.9262 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9545 1.4209 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.5713 1.7534 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-3.0677 1.8344 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.0234 2.3398 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.4066 2.0073 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-3.3717 2.4074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3117 3.0422 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1614 2.2570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8964 2.3163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9165 -2.2150 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7826 -2.7149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 4 1 0 0 0 0
2 10 1 0 0 0 0
10 11 2 0 0 0 0
11 5 1 0 0 0 0
6 12 2 0 0 0 0
7 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
19 20 1 1 0 0 0
20 21 1 1 0 0 0
22 21 1 1 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
19 25 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
24 28 1 0 0 0 0
29 28 1 0 0 0 0
22 1 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
35 36 1 0 0 0 0
34 37 1 0 0 0 0
33 38 1 0 0 0 0
32 39 1 0 0 0 0
31 29 1 0 0 0 0
16 40 1 0 0 0 0
40 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 44
M SMT 1 OCH3
M SVB 1 44 3.2446 -1.9213
S SKP 8
ID FLIA1AGS0006
KNApSAcK_ID C00010081
NAME Formononetin 7-O-rutinoside
CAS_RN 51351-35-0
FORMULA C28H32O13
EXACTMASS 576.18429111
AVERAGEMASS 576.54588
SMILES O(C(C)1)[C@H](OCC([C@@H]2O)O[C@@H](Oc(c5)ccc(c53)C(C(c(c4)ccc(c4)OC)=CO3)=O)[C@H]([C@H]2O)O)[C@H]([C@@H]([C@H](O)1)O)O
M END