Mol:FLIAAADS0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.0766 -0.9823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0766 -0.9823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0766 -1.6247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0766 -1.6247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5203 -1.9458 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5203 -1.9458 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9640 -1.6247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9640 -1.6247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9640 -0.9823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9640 -0.9823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5203 -0.6611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5203 -0.6611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4077 -1.9458 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4077 -1.9458 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8514 -1.6247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8514 -1.6247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8514 -0.9823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8514 -0.9823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4077 -0.6611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4077 -0.6611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4077 -2.4467 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4077 -2.4467 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6327 -0.6612 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6327 -0.6612 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2749 2.0233 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -2.2749 2.0233 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -2.8805 1.5646 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.8805 1.5646 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.6236 0.9039 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.6236 0.9039 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.6167 0.2665 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.6167 0.2665 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.1534 0.7297 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1534 0.7297 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4584 1.3076 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -2.4584 1.3076 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -2.7849 2.9067 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7849 2.9067 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9153 2.2798 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9153 2.2798 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2590 0.5254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2590 0.5254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3160 -2.5794 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3160 -2.5794 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2510 -2.9067 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2510 -2.9067 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8180 -2.5794 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8180 -2.5794 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8180 -1.9247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8180 -1.9247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2510 -1.5973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2510 -1.5973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3160 -1.9247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3160 -1.9247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2946 -3.1176 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.2946 -3.1176 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.7292 -3.3233 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.7292 -3.3233 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.2787 -3.5685 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2787 -3.5685 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6182 -4.0692 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.6182 -4.0692 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.1836 -3.8635 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.1836 -3.8635 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.6342 -3.6183 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.6342 -3.6183 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.7035 -2.7086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7035 -2.7086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9711 -3.2814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9711 -3.2814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6546 -4.0350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6546 -4.0350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3822 -2.8276 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3822 -2.8276 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5203 -2.5880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5203 -2.5880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7004 -4.5204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7004 -4.5204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8786 -4.5922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8786 -4.5922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9315 1.3334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9315 1.3334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4745 2.2228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4745 2.2228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 13 14 1 1 0 0 0 | + | 13 14 1 1 0 0 0 |
| − | 14 15 1 1 0 0 0 | + | 14 15 1 1 0 0 0 |
| − | 16 15 1 1 0 0 0 | + | 16 15 1 1 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 13 19 1 0 0 0 0 | + | 13 19 1 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 6 16 1 0 0 0 0 | + | 6 16 1 0 0 0 0 |
| − | 22 23 2 0 0 0 0 | + | 22 23 2 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 2 0 0 0 0 | + | 24 25 2 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 2 0 0 0 0 | + | 26 27 2 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 27 8 1 0 0 0 0 | + | 27 8 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 1 0 0 0 | + | 29 30 1 1 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 32 31 1 1 0 0 0 | + | 32 31 1 1 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 28 1 0 0 0 0 | + | 33 28 1 0 0 0 0 |
| − | 28 34 1 0 0 0 0 | + | 28 34 1 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 32 36 1 0 0 0 0 | + | 32 36 1 0 0 0 0 |
| − | 29 37 1 0 0 0 0 | + | 29 37 1 0 0 0 0 |
| − | 37 24 1 0 0 0 0 | + | 37 24 1 0 0 0 0 |
| − | 3 38 1 0 0 0 0 | + | 3 38 1 0 0 0 0 |
| − | 31 39 1 0 0 0 0 | + | 31 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 18 41 1 0 0 0 0 | + | 18 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 41 42 | + | M SAL 2 2 41 42 |
| − | M SBL 2 1 45 | + | M SBL 2 1 45 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 45 -1.9315 1.3334 | + | M SVB 2 45 -1.9315 1.3334 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 39 40 | + | M SAL 1 2 39 40 |
| − | M SBL 1 1 43 | + | M SBL 1 1 43 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 43 2.9183 -1.2878 | + | M SVB 1 43 2.9183 -1.2878 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FLIAAADS0001 | + | ID FLIAAADS0001 |
| − | KNApSAcK_ID C00006350 | + | KNApSAcK_ID C00006350 |
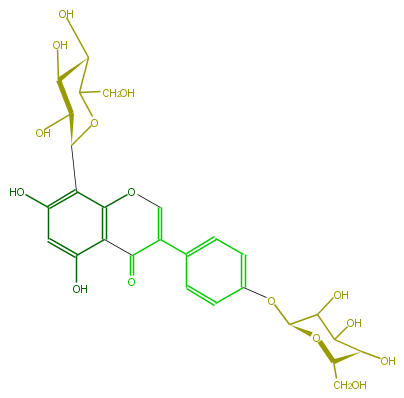
| − | NAME 8-C-Glucopyranosylgenistein 4'-O-glucoside | + | NAME 8-C-Glucopyranosylgenistein 4'-O-glucoside |
| − | CAS_RN 147879-67-2 | + | CAS_RN 147879-67-2 |
| − | FORMULA C27H30O15 | + | FORMULA C27H30O15 |
| − | EXACTMASS 594.15847029 | + | EXACTMASS 594.15847029 |
| − | AVERAGEMASS 594.5181 | + | AVERAGEMASS 594.5181 |
| − | SMILES C(O1)([C@@H]([C@H](O)[C@@H]([C@@H]1c(c(O)5)c(c2c(c5)O)OC=C(c(c3)ccc(O[C@H](O4)C(O)C(O)[C@@H](O)[C@H]4CO)c3)C2=O)O)O)CO | + | SMILES C(O1)([C@@H]([C@H](O)[C@@H]([C@@H]1c(c(O)5)c(c2c(c5)O)OC=C(c(c3)ccc(O[C@H](O4)C(O)C(O)[C@@H](O)[C@H]4CO)c3)C2=O)O)O)CO |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-3.0766 -0.9823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0766 -1.6247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5203 -1.9458 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9640 -1.6247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9640 -0.9823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5203 -0.6611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4077 -1.9458 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8514 -1.6247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8514 -0.9823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4077 -0.6611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4077 -2.4467 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6327 -0.6612 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2749 2.0233 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-2.8805 1.5646 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.6236 0.9039 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.6167 0.2665 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.1534 0.7297 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4584 1.3076 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-2.7849 2.9067 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9153 2.2798 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2590 0.5254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3160 -2.5794 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2510 -2.9067 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8180 -2.5794 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8180 -1.9247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2510 -1.5973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3160 -1.9247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2946 -3.1176 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.7292 -3.3233 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.2787 -3.5685 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6182 -4.0692 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.1836 -3.8635 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.6342 -3.6183 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.7035 -2.7086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9711 -3.2814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6546 -4.0350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3822 -2.8276 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5203 -2.5880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7004 -4.5204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8786 -4.5922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9315 1.3334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4745 2.2228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
1 12 1 0 0 0 0
13 14 1 1 0 0 0
14 15 1 1 0 0 0
16 15 1 1 0 0 0
16 17 1 0 0 0 0
17 18 1 0 0 0 0
18 13 1 0 0 0 0
13 19 1 0 0 0 0
14 20 1 0 0 0 0
15 21 1 0 0 0 0
6 16 1 0 0 0 0
22 23 2 0 0 0 0
23 24 1 0 0 0 0
24 25 2 0 0 0 0
25 26 1 0 0 0 0
26 27 2 0 0 0 0
27 22 1 0 0 0 0
27 8 1 0 0 0 0
28 29 1 0 0 0 0
29 30 1 1 0 0 0
30 31 1 1 0 0 0
32 31 1 1 0 0 0
32 33 1 0 0 0 0
33 28 1 0 0 0 0
28 34 1 0 0 0 0
33 35 1 0 0 0 0
32 36 1 0 0 0 0
29 37 1 0 0 0 0
37 24 1 0 0 0 0
3 38 1 0 0 0 0
31 39 1 0 0 0 0
39 40 1 0 0 0 0
18 41 1 0 0 0 0
41 42 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 41 42
M SBL 2 1 45
M SMT 2 CH2OH
M SVB 2 45 -1.9315 1.3334
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 39 40
M SBL 1 1 43
M SMT 1 CH2OH
M SVB 1 43 2.9183 -1.2878
S SKP 8
ID FLIAAADS0001
KNApSAcK_ID C00006350
NAME 8-C-Glucopyranosylgenistein 4'-O-glucoside
CAS_RN 147879-67-2
FORMULA C27H30O15
EXACTMASS 594.15847029
AVERAGEMASS 594.5181
SMILES C(O1)([C@@H]([C@H](O)[C@@H]([C@@H]1c(c(O)5)c(c2c(c5)O)OC=C(c(c3)ccc(O[C@H](O4)C(O)C(O)[C@@H](O)[C@H]4CO)c3)C2=O)O)O)CO
M END