Mol:FLIFHXGF0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 47 0 0 0 0 0 0 0 0999 V2000 | + | 42 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.1296 2.1674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1296 2.1674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5733 1.8463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5733 1.8463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1196 0.1756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1196 0.1756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4367 0.4968 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4367 0.4968 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9928 0.1758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9928 0.1758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9928 -0.4902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9928 -0.4902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5696 -0.8232 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5696 -0.8232 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1463 -0.4902 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.1463 -0.4902 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.1463 0.1758 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.1463 0.1758 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.5696 0.5087 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5696 0.5087 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1194 -0.4664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1194 -0.4664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4367 -0.7874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4367 -0.7874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5696 -1.4887 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5696 -1.4887 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7227 -0.8229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7227 -0.8229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7227 -1.4596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7227 -1.4596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2740 -1.7779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2740 -1.7779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8254 -1.4596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8254 -1.4596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8254 -0.8229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8254 -0.8229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2740 -0.5046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2740 -0.5046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0700 2.3496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0700 2.3496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3032 1.1251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3032 1.1251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3357 1.1923 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -0.3357 1.1923 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.5969 0.6055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5969 0.6055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2998 0.1758 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2998 0.1758 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7231 0.5087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7231 0.5087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7484 1.4456 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7484 1.4456 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8935 1.0768 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -3.8935 1.0768 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -3.5473 0.6198 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.5473 0.6198 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.0487 0.8137 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.0487 0.8137 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.5290 0.8079 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.5290 0.8079 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.9172 1.1685 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9172 1.1685 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4265 0.9856 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -3.4265 0.9856 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -4.7339 0.8516 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7339 0.8516 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0870 0.5935 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0870 0.5935 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7630 0.3341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7630 0.3341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1463 -1.2327 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1463 -1.2327 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8254 -1.4596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8254 -1.4596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8254 -1.4596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8254 -1.4596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8352 -1.9371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8352 -1.9371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5497 -2.3496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5497 -2.3496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0002 1.7294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0002 1.7294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4127 2.4439 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4127 2.4439 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 7 1 0 0 0 0 | + | 6 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 3 11 1 0 0 0 0 | + | 3 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 6 1 0 0 0 0 | + | 12 6 1 0 0 0 0 |
| − | 7 13 2 0 0 0 0 | + | 7 13 2 0 0 0 0 |
| − | 8 14 1 0 0 0 0 | + | 8 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 19 2 0 0 0 0 | + | 18 19 2 0 0 0 0 |
| − | 19 14 1 0 0 0 0 | + | 19 14 1 0 0 0 0 |
| − | 2 20 2 0 0 0 0 | + | 2 20 2 0 0 0 0 |
| − | 4 21 1 0 0 0 0 | + | 4 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 3 1 0 0 0 0 | + | 23 3 1 0 0 0 0 |
| − | 19 24 1 0 0 0 0 | + | 19 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 9 1 0 0 0 0 | + | 25 9 1 0 0 0 0 |
| − | 22 2 1 0 0 0 0 | + | 22 2 1 0 0 0 0 |
| − | 27 28 1 1 0 0 0 | + | 27 28 1 1 0 0 0 |
| − | 28 29 1 1 0 0 0 | + | 28 29 1 1 0 0 0 |
| − | 30 29 1 1 0 0 0 | + | 30 29 1 1 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 27 1 0 0 0 0 | + | 32 27 1 0 0 0 0 |
| − | 27 33 1 0 0 0 0 | + | 27 33 1 0 0 0 0 |
| − | 28 34 1 0 0 0 0 | + | 28 34 1 0 0 0 0 |
| − | 29 35 1 0 0 0 0 | + | 29 35 1 0 0 0 0 |
| − | 30 26 1 0 0 0 0 | + | 30 26 1 0 0 0 0 |
| − | 26 1 1 0 0 0 0 | + | 26 1 1 0 0 0 0 |
| − | 8 36 1 0 0 0 0 | + | 8 36 1 0 0 0 0 |
| − | 17 37 1 0 0 0 0 | + | 17 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 16 39 1 0 0 0 0 | + | 16 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 32 41 1 0 0 0 0 | + | 32 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 41 42 | + | M SAL 3 2 41 42 |
| − | M SBL 3 1 46 | + | M SBL 3 1 46 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SVB 3 46 -3.6321 1.9022 | + | M SVB 3 46 -3.6321 1.9022 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 39 40 | + | M SAL 2 2 39 40 |
| − | M SBL 2 1 44 | + | M SBL 2 1 44 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 44 2.8352 -1.9371 | + | M SVB 2 44 2.8352 -1.9371 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 37 38 | + | M SAL 1 2 37 38 |
| − | M SBL 1 1 42 | + | M SBL 1 1 42 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 42 4.0195 -1.6658 | + | M SVB 1 42 4.0195 -1.6658 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FLIFHXGF0001 | + | ID FLIFHXGF0001 |
| − | KNApSAcK_ID C00010181 | + | KNApSAcK_ID C00010181 |
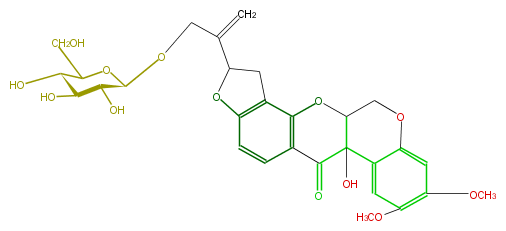
| − | NAME Dalbinol O-glucoside;Dalbin | + | NAME Dalbinol O-glucoside;Dalbin |
| − | CAS_RN 68401-03-6 | + | CAS_RN 68401-03-6 |
| − | FORMULA C29H32O13 | + | FORMULA C29H32O13 |
| − | EXACTMASS 588.18429111 | + | EXACTMASS 588.18429111 |
| − | AVERAGEMASS 588.5565799999999 | + | AVERAGEMASS 588.5565799999999 |
| − | SMILES C(O)C(O1)[C@@H]([C@H](O)[C@H](O)[C@@H]1OCC(=C)C(O2)Cc(c63)c(ccc3C(=O)C(O)(C(O6)4)c(c5)c(cc(OC)c(OC)5)OC4)2)O | + | SMILES C(O)C(O1)[C@@H]([C@H](O)[C@H](O)[C@@H]1OCC(=C)C(O2)Cc(c63)c(ccc3C(=O)C(O)(C(O6)4)c(c5)c(cc(OC)c(OC)5)OC4)2)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 47 0 0 0 0 0 0 0 0999 V2000
-1.1296 2.1674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5733 1.8463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1196 0.1756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4367 0.4968 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9928 0.1758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9928 -0.4902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5696 -0.8232 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1463 -0.4902 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.1463 0.1758 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.5696 0.5087 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1194 -0.4664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4367 -0.7874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5696 -1.4887 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7227 -0.8229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7227 -1.4596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2740 -1.7779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8254 -1.4596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8254 -0.8229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2740 -0.5046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0700 2.3496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3032 1.1251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3357 1.1923 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.5969 0.6055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2998 0.1758 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7231 0.5087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7484 1.4456 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8935 1.0768 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-3.5473 0.6198 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.0487 0.8137 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.5290 0.8079 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.9172 1.1685 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4265 0.9856 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-4.7339 0.8516 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0870 0.5935 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7630 0.3341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1463 -1.2327 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8254 -1.4596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.8254 -1.4596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8352 -1.9371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5497 -2.3496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0002 1.7294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4127 2.4439 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
3 11 1 0 0 0 0
11 12 2 0 0 0 0
12 6 1 0 0 0 0
7 13 2 0 0 0 0
8 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 18 1 0 0 0 0
18 19 2 0 0 0 0
19 14 1 0 0 0 0
2 20 2 0 0 0 0
4 21 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 0 0 0 0
23 3 1 0 0 0 0
19 24 1 0 0 0 0
24 25 1 0 0 0 0
25 9 1 0 0 0 0
22 2 1 0 0 0 0
27 28 1 1 0 0 0
28 29 1 1 0 0 0
30 29 1 1 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
32 27 1 0 0 0 0
27 33 1 0 0 0 0
28 34 1 0 0 0 0
29 35 1 0 0 0 0
30 26 1 0 0 0 0
26 1 1 0 0 0 0
8 36 1 0 0 0 0
17 37 1 0 0 0 0
37 38 1 0 0 0 0
16 39 1 0 0 0 0
39 40 1 0 0 0 0
32 41 1 0 0 0 0
41 42 1 0 0 0 0
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 41 42
M SBL 3 1 46
M SMT 3 CH2OH
M SVB 3 46 -3.6321 1.9022
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 39 40
M SBL 2 1 44
M SMT 2 OCH3
M SVB 2 44 2.8352 -1.9371
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 37 38
M SBL 1 1 42
M SMT 1 OCH3
M SVB 1 42 4.0195 -1.6658
S SKP 8
ID FLIFHXGF0001
KNApSAcK_ID C00010181
NAME Dalbinol O-glucoside;Dalbin
CAS_RN 68401-03-6
FORMULA C29H32O13
EXACTMASS 588.18429111
AVERAGEMASS 588.5565799999999
SMILES C(O)C(O1)[C@@H]([C@H](O)[C@H](O)[C@@H]1OCC(=C)C(O2)Cc(c63)c(ccc3C(=O)C(O)(C(O6)4)c(c5)c(cc(OC)c(OC)5)OC4)2)O
M END