Mol:FLNACCGS0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 33 36 0 0 0 0 0 0 0 0999 V2000 | + | 33 36 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.5385 1.4802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5385 1.4802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0178 1.8014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0178 1.8014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5739 1.4804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5739 1.4804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5739 0.8144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5739 0.8144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1507 0.4814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1507 0.4814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7274 0.8144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7274 0.8144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7274 1.4804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7274 1.4804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1507 1.8133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1507 1.8133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5385 0.8382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5385 0.8382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0178 0.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0178 0.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1507 -0.1841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1507 -0.1841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5977 -0.5033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5977 -0.5033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5977 -1.1418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5977 -1.1418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1507 -1.4610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1507 -1.4610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7036 -1.1418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7036 -1.1418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7036 -0.5033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7036 -0.5033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3037 1.8131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3037 1.8131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1507 -2.0990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1507 -2.0990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2561 -1.4608 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2561 -1.4608 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0163 -0.1190 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0163 -0.1190 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9033 -0.5341 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -1.9033 -0.5341 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -1.5451 -1.0070 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.5451 -1.0070 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.0292 -0.8064 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.0292 -0.8064 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.5315 -0.8010 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.5315 -0.8010 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.8932 -0.4393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8932 -0.4393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4201 -0.6285 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -1.4201 -0.6285 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -2.3638 -0.4939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3638 -0.4939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8832 -1.4521 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8832 -1.4521 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7337 -1.3025 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7337 -1.3025 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8957 2.0990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8957 2.0990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3956 2.9651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3956 2.9651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6924 0.1035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6924 0.1035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6784 0.2702 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6784 0.2702 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 7 1 0 0 0 0 | + | 6 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 3 1 0 0 0 0 | + | 8 3 1 0 0 0 0 |
| − | 1 9 1 0 0 0 0 | + | 1 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 4 1 0 0 0 0 | + | 10 4 1 0 0 0 0 |
| − | 5 11 1 0 0 0 0 | + | 5 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 20 24 1 0 0 0 0 | + | 20 24 1 0 0 0 0 |
| − | 10 20 1 0 0 0 0 | + | 10 20 1 0 0 0 0 |
| − | 1 30 1 0 0 0 0 | + | 1 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 26 32 1 0 0 0 0 | + | 26 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 32 33 | + | M SAL 2 2 32 33 |
| − | M SBL 2 1 35 | + | M SBL 2 1 35 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 35 -1.6924 0.1035 | + | M SVB 2 35 -1.6924 0.1035 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 30 31 | + | M SAL 1 2 30 31 |
| − | M SBL 1 1 33 | + | M SBL 1 1 33 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 33 -0.8957 2.099 | + | M SVB 1 33 -0.8957 2.099 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FLNACCGS0001 | + | ID FLNACCGS0001 |
| − | KNApSAcK_ID C00010235 | + | KNApSAcK_ID C00010235 |
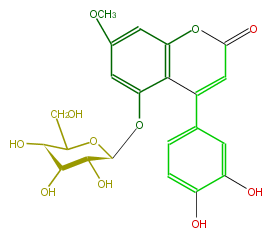
| − | NAME 5,3',4'-Trihydroxy-7-methoxy-4-phenylcoumarin 5-O-glucoside | + | NAME 5,3',4'-Trihydroxy-7-methoxy-4-phenylcoumarin 5-O-glucoside |
| − | CAS_RN 116310-58-8 | + | CAS_RN 116310-58-8 |
| − | FORMULA C22H22O11 | + | FORMULA C22H22O11 |
| − | EXACTMASS 462.116211546 | + | EXACTMASS 462.116211546 |
| − | AVERAGEMASS 462.40348000000006 | + | AVERAGEMASS 462.40348000000006 |
| − | SMILES c(c(O[C@@H]([C@@H](O)4)OC(CO)[C@@H]([C@@H]4O)O)1)c(OC)cc(O3)c1C(=CC(=O)3)c(c2)ccc(c2O)O | + | SMILES c(c(O[C@@H]([C@@H](O)4)OC(CO)[C@@H]([C@@H]4O)O)1)c(OC)cc(O3)c1C(=CC(=O)3)c(c2)ccc(c2O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
33 36 0 0 0 0 0 0 0 0999 V2000
-0.5385 1.4802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0178 1.8014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5739 1.4804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5739 0.8144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1507 0.4814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7274 0.8144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7274 1.4804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1507 1.8133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5385 0.8382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0178 0.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1507 -0.1841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5977 -0.5033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5977 -1.1418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1507 -1.4610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7036 -1.1418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7036 -0.5033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3037 1.8131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1507 -2.0990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2561 -1.4608 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0163 -0.1190 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9033 -0.5341 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-1.5451 -1.0070 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.0292 -0.8064 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.5315 -0.8010 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.8932 -0.4393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4201 -0.6285 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-2.3638 -0.4939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8832 -1.4521 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7337 -1.3025 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8957 2.0990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3956 2.9651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6924 0.1035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6784 0.2702 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 7 1 0 0 0 0
7 8 1 0 0 0 0
8 3 1 0 0 0 0
1 9 1 0 0 0 0
9 10 2 0 0 0 0
10 4 1 0 0 0 0
5 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
14 18 1 0 0 0 0
15 19 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
20 24 1 0 0 0 0
10 20 1 0 0 0 0
1 30 1 0 0 0 0
30 31 1 0 0 0 0
26 32 1 0 0 0 0
32 33 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 32 33
M SBL 2 1 35
M SMT 2 CH2OH
M SVB 2 35 -1.6924 0.1035
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 30 31
M SBL 1 1 33
M SMT 1 OCH3
M SVB 1 33 -0.8957 2.099
S SKP 8
ID FLNACCGS0001
KNApSAcK_ID C00010235
NAME 5,3',4'-Trihydroxy-7-methoxy-4-phenylcoumarin 5-O-glucoside
CAS_RN 116310-58-8
FORMULA C22H22O11
EXACTMASS 462.116211546
AVERAGEMASS 462.40348000000006
SMILES c(c(O[C@@H]([C@@H](O)4)OC(CO)[C@@H]([C@@H]4O)O)1)c(OC)cc(O3)c1C(=CC(=O)3)c(c2)ccc(c2O)O
M END