Aritalab:Lecture/Biochem/Modularity/NF
Contents |
MAPK カスケードと負のフィードバック
ここでは Cell Designer ソフトウェアを用いて、MAPK カスケードのシミュレーションを実感することを目標としています。 Cell Designer ソフトウェアのデータベースBioModelsから、 MAPK を検索してBIOMD0000000010を選択してください。
|
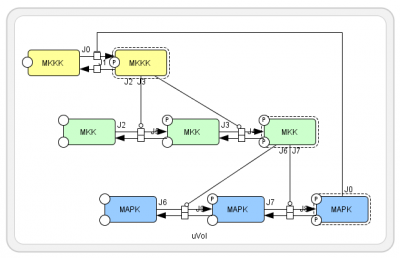
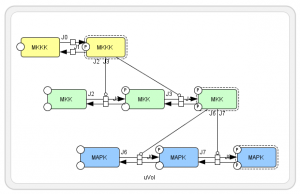
Mitogen-activated Protein Kinase (MAPK) は全身の細胞で発現し、細胞外のシグナルを核内に伝える代表的なシグナル伝達経路です。 MAPK とは総称で、代表的なものにERK1/2があります。ここでは MAPK が MAPキナーゼキナーゼ (MAPKK) により 2 段階のリン酸化を受け、MAPKK はMAPKKキナーゼにより 2 段階のリン酸化を受けるモデルになっています。最終的にできるMAPK二リン酸は、MKKK を抑制しています。 左図の右下から左上に戻ってくる経路 (抑制する反応を┴型で表現) が、抑制効果を表しています。 このサイクルには、一つだけ「抑制」の効果があるので、負のフィードバックループになっています。 |
|
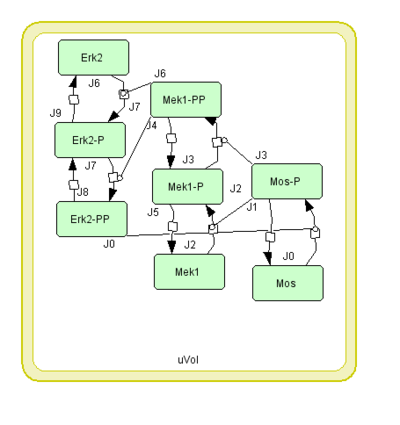
BioModelsからダウンロードしたモデルが上のモデルと同じかどうかは一見わかりにくいですが、エディタでタンパク質の位置関係を整理すると、同じモデルであることがわかります。
という対応関係です。最後のMAPKによるフィードバック部分は MKKK / ((1 + pow(MAPK_PP / Ki, n)) * (K1 + MKKK)) というヒル式になっています。 |
|
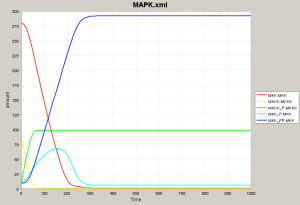
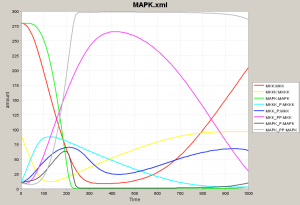
とりあえず経路全体をシミュレーションしてみます(Simulationパネル)。8種のタンパク質が複雑な動きをすることがわかります。 初期値をみると Mos, Mek1, Erk2 がそれぞれ 90, 280, 280 になっています。(リン酸化されていない初期状態。) シミュレーション結果のグラフでは、黄色、赤、緑色の線がそれぞれ 90, 280, 280 の値からスタートしているのがわかります。 残りの初期値はすべて 10 になっていて、全てリン酸化されたタンパク質を表現しています。Mek1, Erk2 は2段階のリン酸化がおこなわれます。 |
|
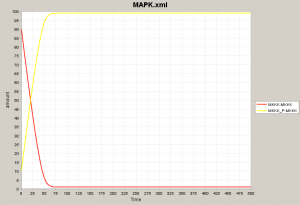
ここでフィードバックに当たる部分のヒル式を MKKK / (K1 + MKKK) 形に修正し、フィードバックをなくします。ループがないため、MosとMos-P(リン酸化された状態)が平衡状態、つまり量の変動がない状態に落ち着きます。 左図で黄色が Mos-P, 赤色が Mos です。全ての Mos がリン酸化されてそのまま動かないことがわかります。 Mos とMos-Pはそれぞれ 90, 10 が初期値だったので合計した 100 が最終的な Mos-P の量になっています。 |
|
つぎにシミュレーションパネルのチェックボックスを利用して、Mos, Mek1だけの表示にしてみます。フィードバックループがないため、やはり量の変動がない状態に落ち着くはずです。 |
|
Mosとそのリン酸化体、Mek1とそのリン酸化体はいずれも相補的な挙動をみせています。二段階リン酸化を経る場合、一段階目のリン酸化体は量が大きく増えない点にも注意しましょう。 |
|
ここまでの知見はカスケードが何段階になっても同じです。フィードバックがない状態では、それぞれのタンパク質がリン酸化されておわります。 |
|
フィードバックがない状態で8種類のタンパク質を観測すると
という3組の動きがどれも似て、いずれも相補的に動いていることがわかります。(ちょっとわかりにくいですが色を確認してください。) 量が変化しはじめる時間をみると、その順序は Mos → Mek1 → Erk2 です。Mek2の薄いグレーだけは増加のスピードが速くマゼンタを追い抜いて量が増えますが、増加し始めるタイミングはマゼンタより遅いことに注目してください。 |
|
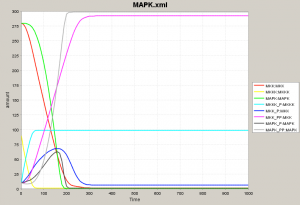
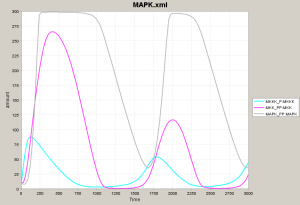
ここで最初のネットワークに戻って、フィードバックを考慮した状態でシミュレーションをやってみましょう。 黄色と水色、赤とマゼンタ、緑と薄いグレーが相補的な動きを見せるところは一緒です。 ネットワーク図における各タンパク質をモジュールと考え、代表タンパク質 (Mos-P, Mek1-PP, Erk2-PP) だけを表示させてみます。 この3者がネットワークの挙動を示す鍵となっています。 薄いグレー色が上がってくるにつれて量が増えていた水色 (Mos-P) が減ってくることに注意してください。 水色が減るということは、これと相補的な挙動をする黄色 (Mos) が増えるということで、シミュレーションの初期に近い状態に戻っていきます。 |
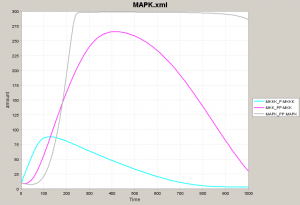
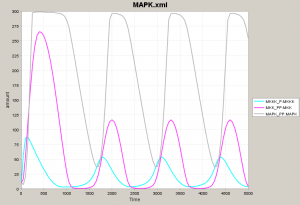
ここまで読んできた人は、最初のシミュレーション結果を複雑で難しいとは思わなくなります。なぜなら、3つのモジュールに分けて考えればよく、水色、マゼンタ、薄いグレーの挙動だけ把握すればあとは補えることがわかったからです。 下の図で左が「難しく見える」シミュレーション結果です。このうち、3モジュールに注目すると真ん中のようになります。結局、3つのタンパク質グループ(ネットワーク図における黄色、黄緑、青の3種)が順番に量を増加させ、順番に量を減少させるだけです。 ですから、時間軸を長くとると右図のようになり、振動していることがわかります。
|
|
|
まとめ
- 負のフィードバックは、振動現象を実現する。